⚠️ 以下所有内容总结都来自于 大语言模型的能力,如有错误,仅供参考,谨慎使用
🔴 请注意:千万不要用于严肃的学术场景,只能用于论文阅读前的初筛!
💗 如果您觉得我们的项目对您有帮助 ChatPaperFree ,还请您给我们一些鼓励!⭐️ HuggingFace免费体验
2025-03-20 更新
Scale-Aware Contrastive Reverse Distillation for Unsupervised Medical Anomaly Detection
Authors:Chunlei Li, Yilei Shi, Jingliang Hu, Xiao Xiang Zhu, Lichao Mou
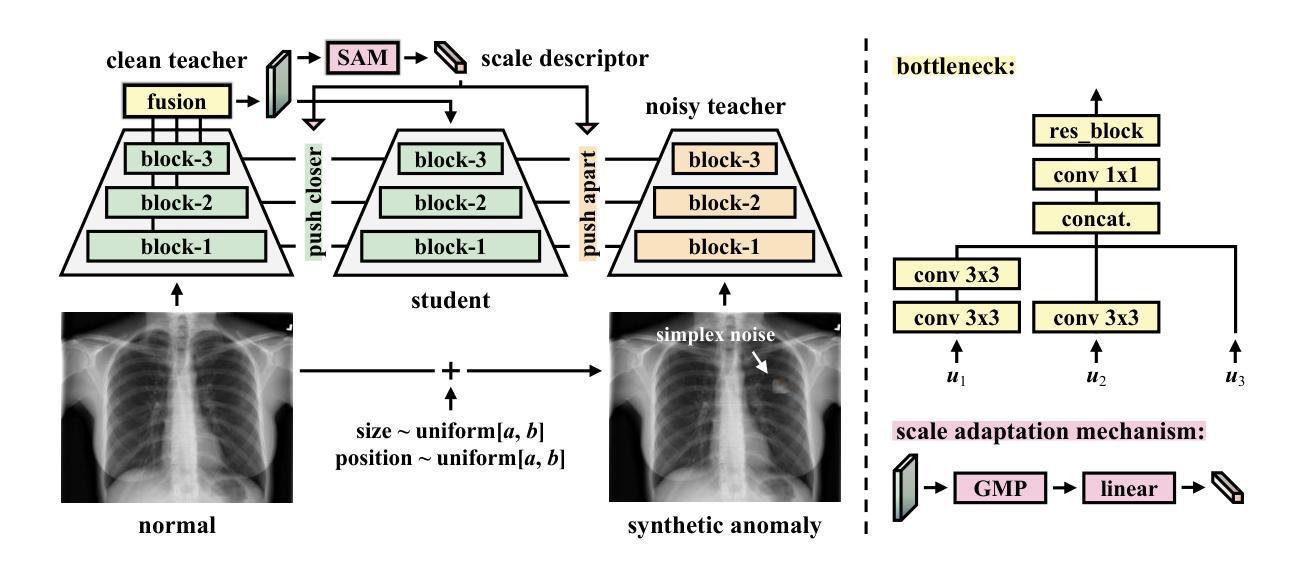
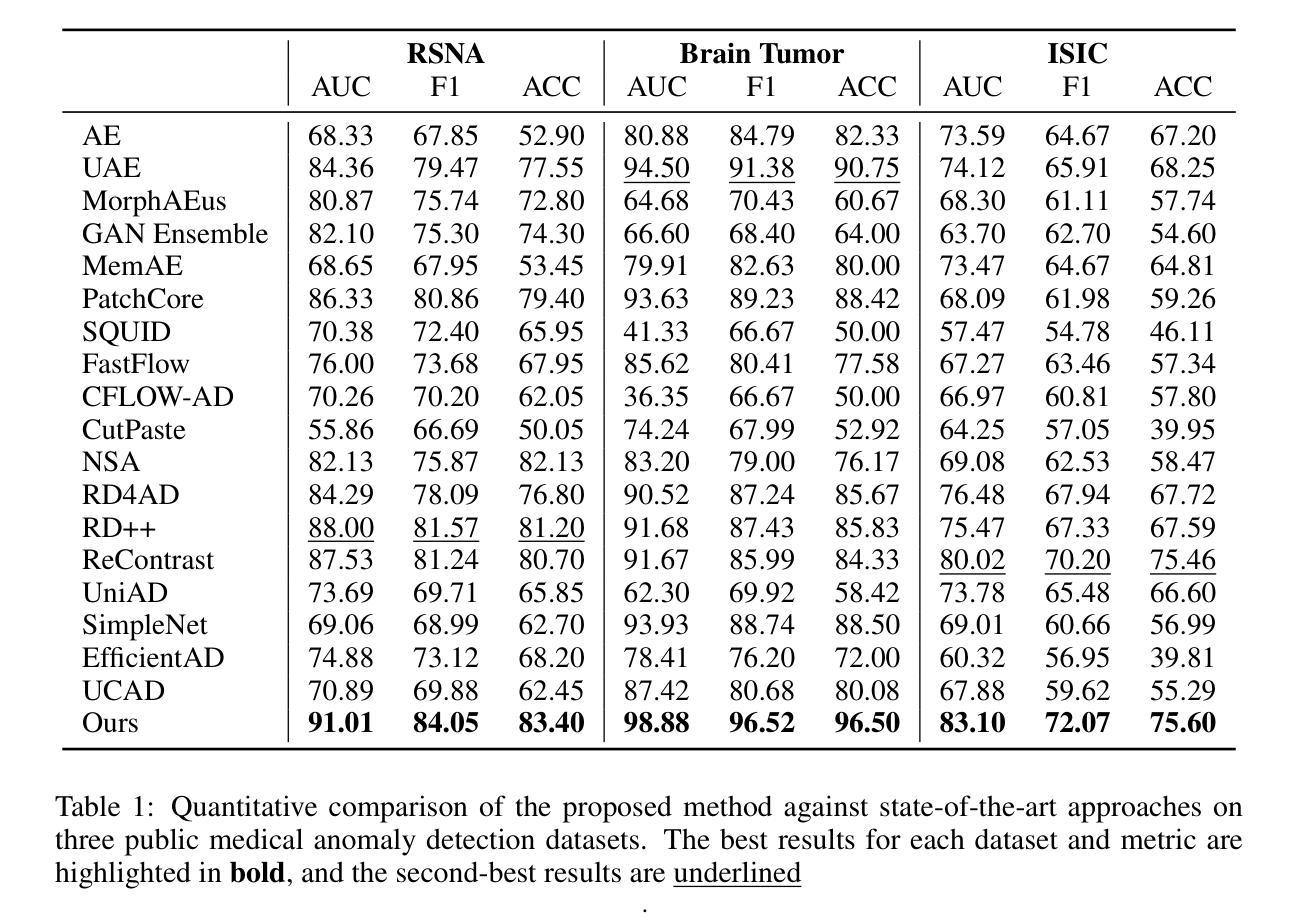
Unsupervised anomaly detection using deep learning has garnered significant research attention due to its broad applicability, particularly in medical imaging where labeled anomalous data are scarce. While earlier approaches leverage generative models like autoencoders and generative adversarial networks (GANs), they often fall short due to overgeneralization. Recent methods explore various strategies, including memory banks, normalizing flows, self-supervised learning, and knowledge distillation, to enhance discrimination. Among these, knowledge distillation, particularly reverse distillation, has shown promise. Following this paradigm, we propose a novel scale-aware contrastive reverse distillation model that addresses two key limitations of existing reverse distillation methods: insufficient feature discriminability and inability to handle anomaly scale variations. Specifically, we introduce a contrastive student-teacher learning approach to derive more discriminative representations by generating and exploring out-of-normal distributions. Further, we design a scale adaptation mechanism to softly weight contrastive distillation losses at different scales to account for the scale variation issue. Extensive experiments on benchmark datasets demonstrate state-of-the-art performance, validating the efficacy of the proposed method. Code is available at https://github.com/MedAITech/SCRD4AD.
基于深度学习的无监督异常检测因其广泛的应用性而受到了研究者的广泛关注,特别是在医学影像领域,异常数据标注稀少。尽管早期的方法利用生成模型,如自动编码器和生成对抗网络(GANs),但由于过度泛化,它们通常无法达到预期效果。最近的方法探索了各种策略,包括内存银行、归一化流、自监督学习和知识蒸馏,以增强鉴别能力。其中,知识蒸馏,特别是反向蒸馏显示出良好的前景。遵循这一范式,我们提出了一种新颖的尺度感知对比反向蒸馏模型,该模型解决了现有反向蒸馏方法的两个关键局限性:特征鉴别能力不足以及处理异常尺度变化的能力不足。具体来说,我们引入了一种对比学生-教师学习方法,通过生成和探索非正常分布来得出更具鉴别力的表示。此外,我们设计了一种尺度适应机制,以不同尺度上轻柔地加权对比蒸馏损失,以解决尺度变化问题。在基准数据集上的大量实验证明了所提出方法的卓越性能,验证了其有效性。代码可在 https://github.com/MedAITech/SCRD4AD 找到。
论文及项目相关链接
PDF ICLR 2025
Summary
本文介绍了基于深度学习的无监督异常检测在医疗影像领域的应用。针对现有方法存在的问题,如过度泛化和无法处理异常尺度变化等,提出了一种新的基于对比反向蒸馏模型的异常检测方法。该方法引入对比性学生-教师学习机制,生成并探索异常分布,同时设计了一种尺度适应机制来处理不同尺度的对比蒸馏损失。在基准数据集上的实验证明了该方法的有效性。
Key Takeaways
- 无监督异常检测在医疗影像等领域受到广泛关注,尤其是因缺乏标注异常数据的情况下。
- 现有方法如利用生成模型(如自编码器、生成对抗网络等)存在过度泛化的问题。
- 知识蒸馏,特别是反向蒸馏技术在异常检测中显示出潜力。
- 本文提出了一种新的基于对比反向蒸馏的异常检测方法,解决了现有方法的局限性。
- 引入对比性学生-教师学习机制,旨在生成并探索更具区分性的表示。
- 设计了一种尺度适应机制,以处理不同尺度的对比蒸馏损失,应对异常尺度的变化。
点此查看论文截图

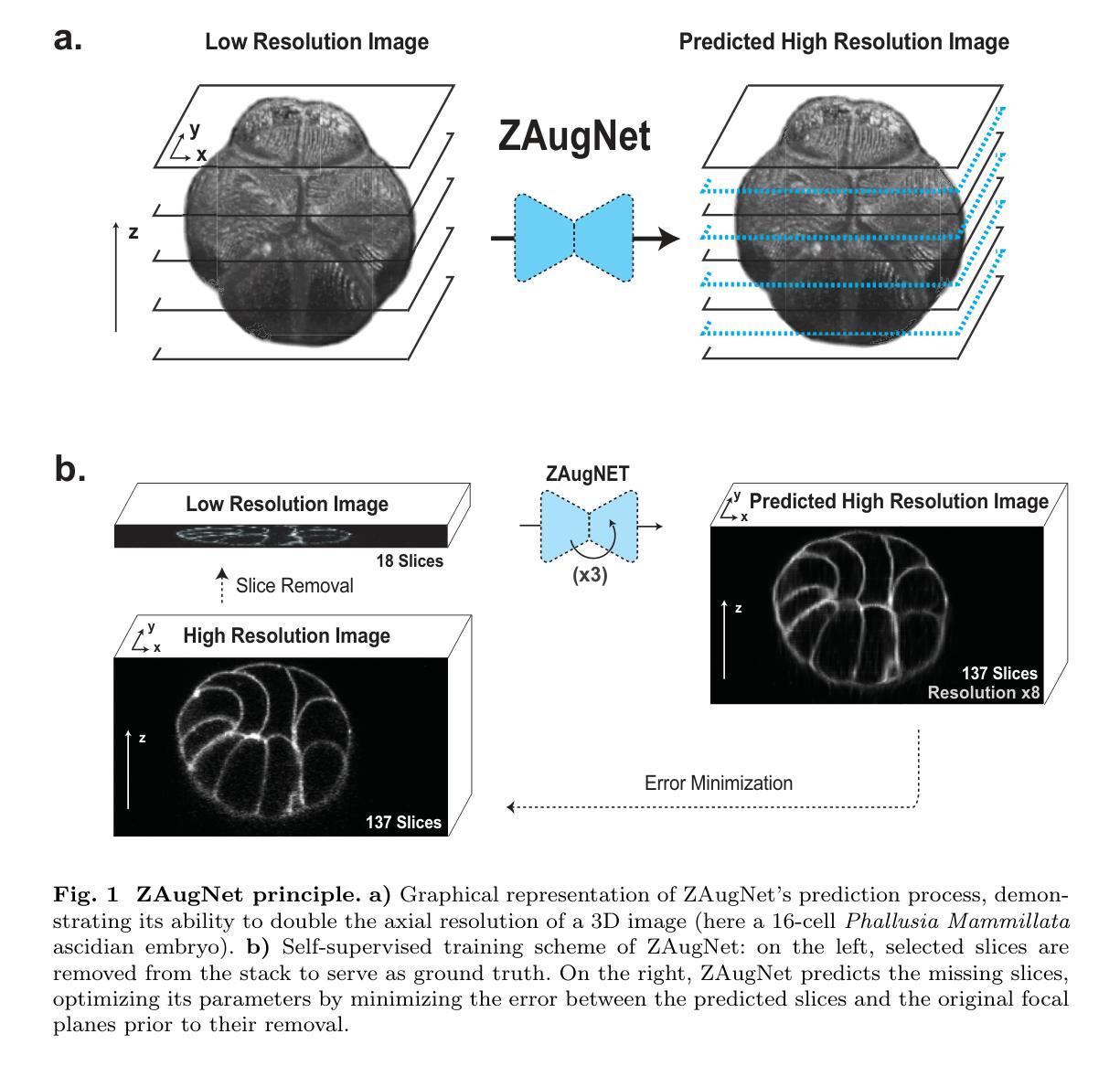
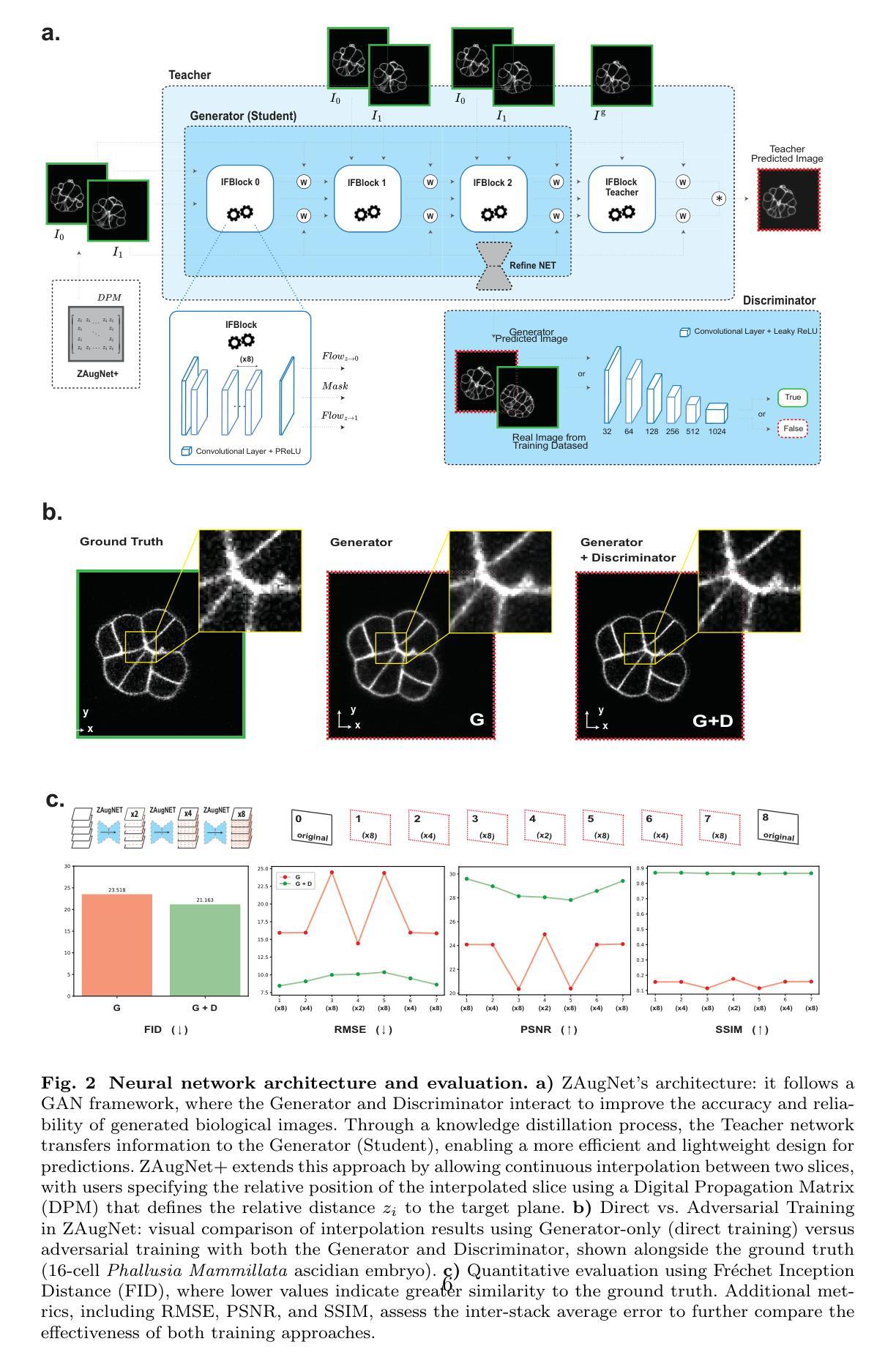
Self-Supervised Z-Slice Augmentation for 3D Bio-Imaging via Knowledge Distillation
Authors:Alessandro Pasqui, Sajjad Mahdavi, Benoit Vianay, Alexandra Colin, Alex McDougall, Rémi Dumollard, Yekaterina A. Miroshnikova, Elsa Labrune, Hervé Turlier
Three-dimensional biological microscopy has significantly advanced our understanding of complex biological structures. However, limitations due to microscopy techniques, sample properties or phototoxicity often result in poor z-resolution, hindering accurate cellular measurements. Here, we introduce ZAugNet, a fast, accurate, and self-supervised deep learning method for enhancing z-resolution in biological images. By performing nonlinear interpolation between consecutive slices, ZAugNet effectively doubles resolution with each iteration. Compared on several microscopy modalities and biological objects, it outperforms competing methods on most metrics. Our method leverages a generative adversarial network (GAN) architecture combined with knowledge distillation to maximize prediction speed without compromising accuracy. We also developed ZAugNet+, an extended version enabling continuous interpolation at arbitrary distances, making it particularly useful for datasets with nonuniform slice spacing. Both ZAugNet and ZAugNet+ provide high-performance, scalable z-slice augmentation solutions for large-scale 3D imaging. They are available as open-source frameworks in PyTorch, with an intuitive Colab notebook interface for easy access by the scientific community.
三维生物显微镜已经极大地推动了我们对于复杂生物结构的理解。然而,由于显微镜技术、样本特性或光毒性等限制因素,常常导致z轴分辨率不佳,阻碍了精确的细胞测量。在这里,我们引入了ZAugNet,这是一种快速、精确、自我监督的深度学习方法,用于提高生物图像中的z轴分辨率。通过连续切片之间进行非线性插值运算,ZAugNet每次迭代都能有效地将分辨率提高一倍。在多种显微镜技术和生物对象上进行了比较,它在大多数指标上的表现都优于其他方法。我们的方法利用生成对抗网络(GAN)架构,结合知识蒸馏技术,以最大化预测速度而不损害准确性。我们还开发了ZAugNet+这一扩展版本,可实现任意距离的连续插值,使其对于切片间距不均匀的数据集特别有用。ZAugNet和ZAugNet+均提供高性能、可扩展的z轴切片增强解决方案,适用于大规模三维成像。它们可作为PyTorch中的开源框架使用,并提供直观的Colab笔记本界面,方便科学界轻松访问。
论文及项目相关链接
PDF 25 pages, 5 figures, 1 table
Summary
三维生物显微镜技术对复杂生物结构的研究有着巨大推动作用,但由于显微镜技术、样本特性或光毒性等限制,z分辨率往往不佳,影响细胞测量的准确性。本文介绍了一种快速、精准、自我监督的深度学习方法ZAugNet,它提高了生物图像的z分辨率。通过连续切片间的非线性插值,ZAugNet每次迭代都能有效地将分辨率提高一倍。在不同显微镜模式和生物对象上的比较显示,它在大多数指标上都优于其他方法。该方法采用生成对抗网络(GAN)架构结合知识蒸馏技术,以最大化预测速度而不损失准确性。此外,还开发了ZAugNet+版本,支持任意距离的连续插值,适用于非均匀切片间距的数据集。ZAugNet和ZAugNet+为大规模三维成像提供了高性能、可扩展的z切片增强解决方案,并以PyTorch的开源框架形式提供,配有直观的Colab笔记本界面,方便科研界使用。
Key Takeaways
- ZAugNet是一种深度学习技术,旨在提高三维生物显微镜图像的z分辨率。
- 通过非线性插值连续切片,ZAugNet可以每次迭代将图像分辨率提高一倍。
- ZAugNet在多种显微镜模式和生物对象上的性能优于其他方法。
- ZAugNet采用生成对抗网络(GAN)和知识蒸馏技术,以提高预测速度和准确性。
- ZAugNet+是ZAugNet的扩展版本,支持任意距离的连续插值,适用于非均匀切片间距的数据。
- ZAugNet和ZAugNet+为大规模三维成像提供了高性能、可扩展的z切片增强解决方案。
点此查看论文截图