⚠️ 以下所有内容总结都来自于 大语言模型的能力,如有错误,仅供参考,谨慎使用
🔴 请注意:千万不要用于严肃的学术场景,只能用于论文阅读前的初筛!
💗 如果您觉得我们的项目对您有帮助 ChatPaperFree ,还请您给我们一些鼓励!⭐️ HuggingFace免费体验
2025-06-04 更新
NMCSE: Noise-Robust Multi-Modal Coupling Signal Estimation Method via Optimal Transport for Cardiovascular Disease Detection
Authors:Peihong Zhang, Zhixin Li, Rui Sang, Yuxuan Liu, Yiqiang Cai, Yizhou Tan, Shengchen Li
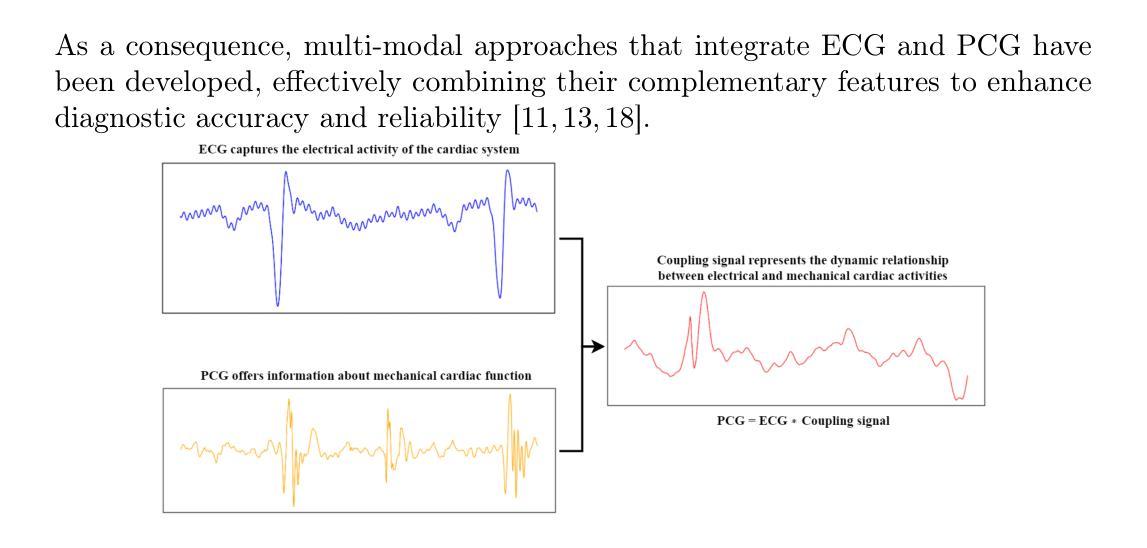
Electrocardiogram (ECG) and Phonocardiogram (PCG) signals are linked by a latent coupling signal representing the electrical-to-mechanical cardiac transformation. While valuable for cardiovascular disease (CVD) detection, this coupling signal is traditionally estimated using deconvolution methods that amplify noise, limiting clinical utility. In this paper, we propose Noise-Robust Multi-Modal Coupling Signal Estimation (NMCSE), which reformulates the problem as distribution matching via optimal transport theory. By jointly optimizing amplitude and temporal alignment, NMCSE mitigates noise amplification without additional preprocessing. Integrated with our Temporal-Spatial Feature Extraction network, NMCSE enables robust multi-modal CVD detection. Experiments on the PhysioNet 2016 dataset with realistic hospital noise demonstrate that NMCSE reduces estimation errors by approximately 30% in Mean Squared Error while maintaining higher Pearson Correlation Coefficients across all tested signal-to-noise ratios. Our approach achieves 97.38% accuracy and 0.98 AUC in CVD detection, outperforming state-of-the-art methods and demonstrating robust performance for real-world clinical applications.
心电图(ECG)和心音图(PCG)信号通过代表电-机械心脏转换的潜在耦合信号相关联。虽然这种耦合信号对于检测心血管疾病(CVD)很有价值,但传统上是通过反卷积方法来估计的,这会增加噪声,限制了其在临床上的实用价值。在本文中,我们提出了噪声鲁棒的跨模态耦合信号估计(NMCSE),它将问题重新定义为通过最优传输理论进行分布匹配。通过联合优化幅度和时间对齐,NMCSE可以在无需额外预处理的情况下减轻噪声放大。结合我们的时空特征提取网络,NMCSE可实现稳健的跨模态CVD检测。在具有现实医院噪声的PhysioNet 2016数据集上的实验表明,在均方误差方面,NMCSE将估计误差降低了约30%,并且在所有测试的信噪比中保持了更高的皮尔逊相关系数。我们的方法在CVD检测中达到了97.38%的准确度和0.98的AUC值,优于最新方法,并展示了在现实临床应用中的稳健性能。
论文及项目相关链接
Summary
本文提出一种噪声鲁棒的跨模态耦合信号估计方法(NMCSE),用于估计心电图(ECG)和心音图(PCG)之间的潜在耦合信号。该方法基于最优传输理论重新构建问题,联合优化振幅和时间对齐,降低了噪声放大问题,并与我们的时空特征提取网络相结合,实现了稳健的多模态心血管疾病(CVD)检测。在PhysioNet 2016数据集上的实验表明,NMCSE在平均误差平方上减少了大约30%的估计误差,同时在所有测试的信号噪声比上都保持了较高的皮尔逊相关系数。在心血管疾病检测中,其准确性达到97.38%,AUC达到0.98,优于现有方法,并在现实的临床应用中表现出稳健性能。
Key Takeaways
- 心电图(ECG)和心音图(PCG)之间的耦合信号代表了心脏的电-机械转换。
- 传统估计耦合信号的方法使用解卷积方法,会放大噪声,限制了其在临床的实用性。
- 本文提出噪声鲁棒的跨模态耦合信号估计方法(NMCSE),通过最优传输理论重新构建问题来解决噪声问题。
- NMCSE联合优化振幅和时间对齐,降低了噪声放大问题,提高了估计的准确性。
- NMCSE与时空特征提取网络结合,实现了多模态心血管疾病检测。
- 在PhysioNet数据集上的实验结果显示,NMCSE显著减少了估计误差,并提高了检测性能。
点此查看论文截图

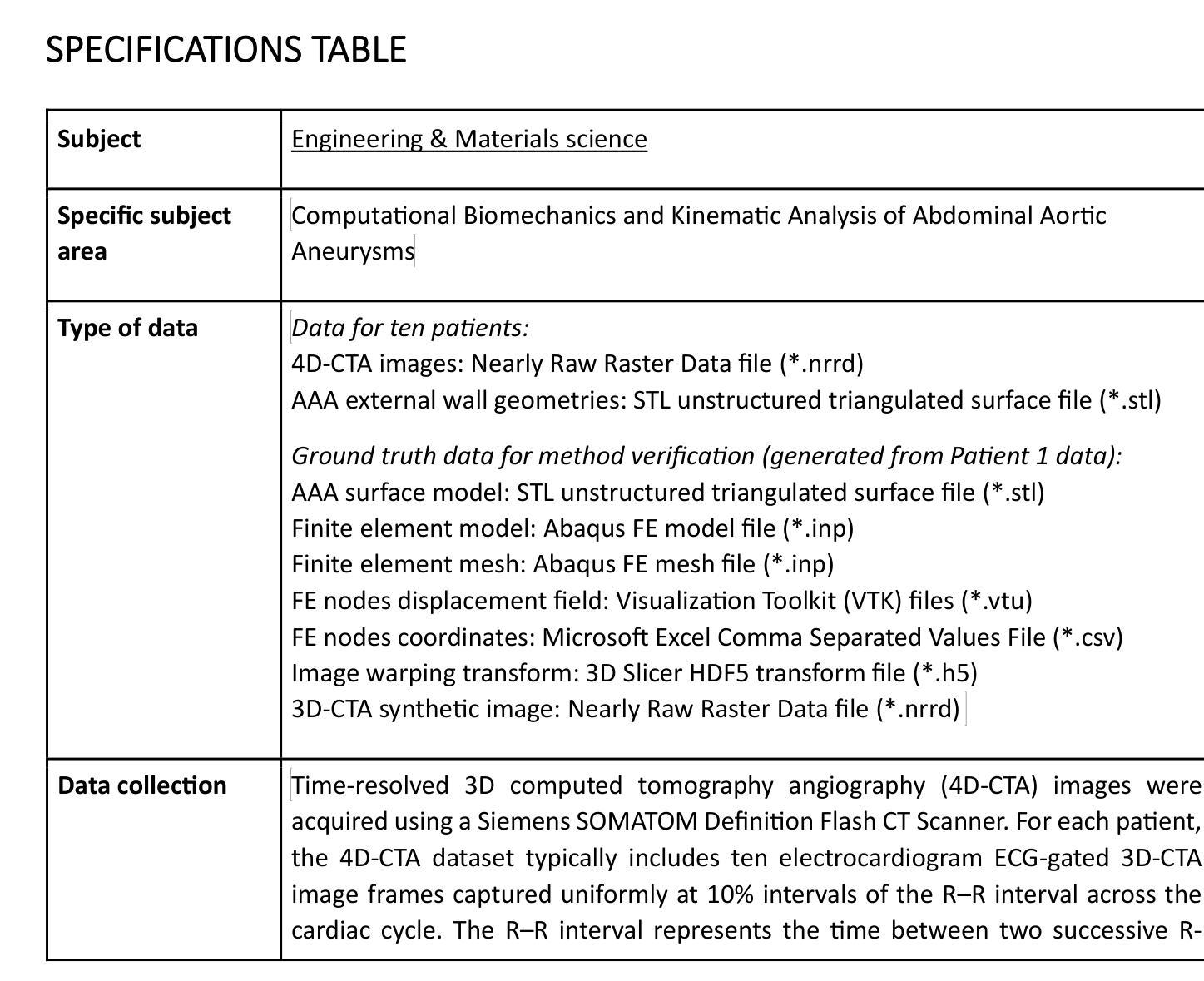
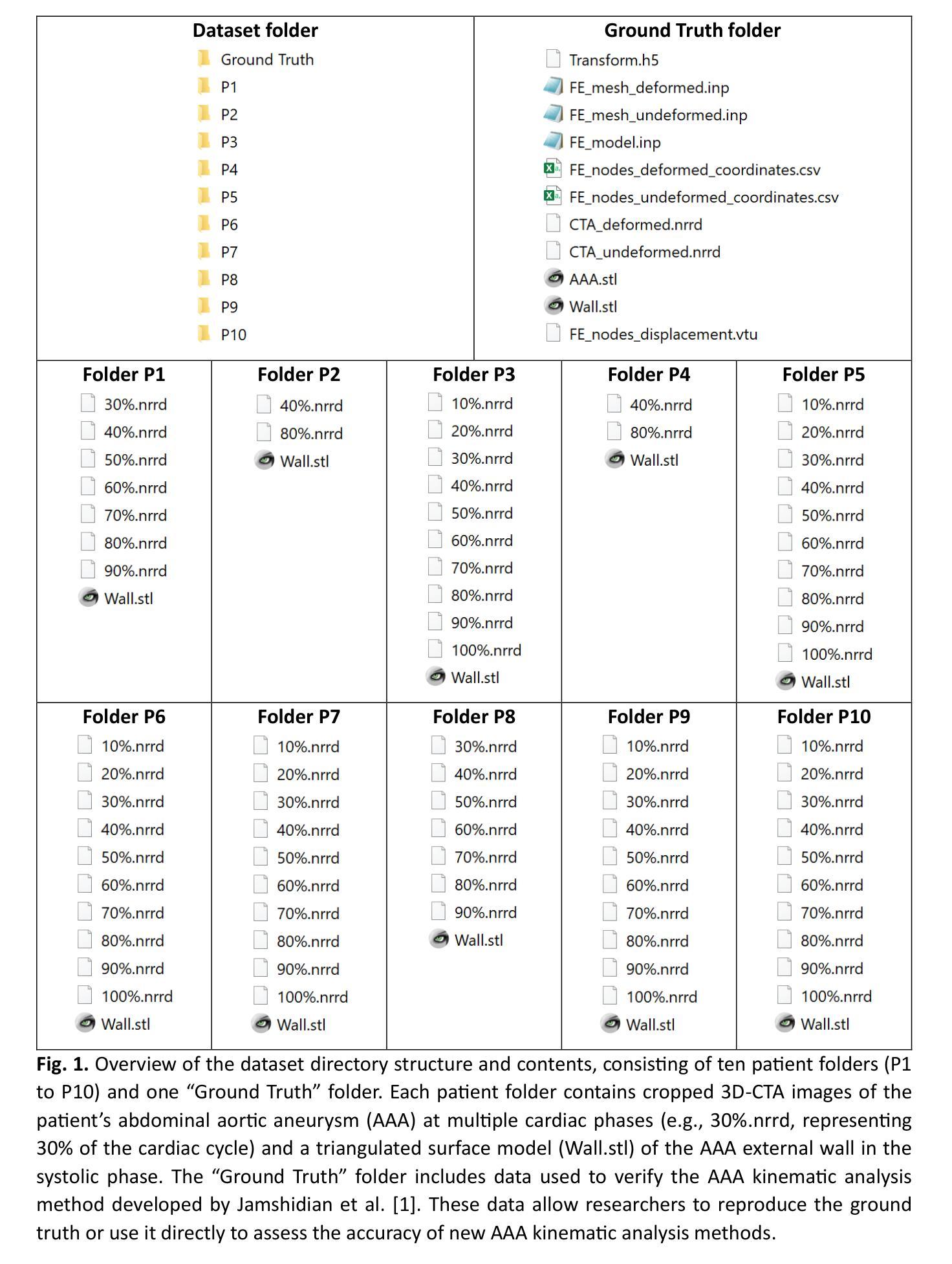
4D-CTA Image and geometry dataset for kinematic analysis of abdominal aortic aneurysms
Authors:Mostafa Jamshidian, Adam Wittek, Saeideh Sekhavat, Farah Alkhatib, Jens Carsten Ritter, Paul M. Parizel, Donatien Le Liepvre, Florian Bernard, Ludovic Minvielle, Antoine Fondanèche, Jane Polce, Christopher Wood, Karol Miller
This article presents a dataset used in the article “Kinematics of Abdominal Aortic Aneurysms”, published in the Journal of Biomechanics. The dataset is publicly available for download from the Zenodo data repository (https://doi.org/10.5281/zenodo.15477710). The dataset includes time-resolved 3D computed tomography angiography (4D-CTA) images of abdominal aortic aneurysm (AAA) captured throughout the cardiac cycle from ten patients diagnosed with AAA, along with ten patient-specific AAA geometries extracted from these images. Typically, the 4D-CTA dataset for each patient contains ten electrocardiogram (ECG)-gated 3D-CTA image frames acquired over a cardiac cycle, capturing both the systolic and diastolic phases of the AAA configuration. For method verification, the dataset also includes synthetic ground truth data generated from Patient 1’s 3D-CTA AAA image in the diastolic phase. The ground truth data includes the patient-specific finite element (FE) biomechanical model and a synthetic systolic 3D-CTA image. The synthetic systolic image was generated by warping Patient 1’s diastolic 3D-CTA image using the realistic displacement field obtained from the AAA biomechanical FE model. The images were acquired at Fiona Stanley Hospital in Western Australia and provided to the researchers at the Intelligent Systems for Medicine Laboratory at The University of Western Australia (ISML-UWA), where image-based AAA kinematic analysis was performed. Our dataset enabled the analysis of AAA wall displacement and strain throughout the cardiac cycle using a non-invasive, in vivo, image registration-based approach. The use of widely adopted, open-source file formats (NRRD for images and STL for geometries) facilitates broad applicability and reusability in AAA biomechanics studies that require patient-specific geometry and information about AAA kinematics during cardiac cycle.
本文介绍了一个在《生物力学杂志》上发表的名为“腹部主动脉瘤的运动学”的文章中使用的数据集。该数据集可从Zenodo数据仓库(https://doi.org/10.5281/zenodo.15477710)公开下载。数据集包含通过心脏周期捕获的腹部主动脉瘤(AAA)的时间分辨三维计算机断层血管造影(4D-CTA)图像,包括10名被诊断为AAA的患者的AAA图像,以及从这些图像中提取的10个患者特定的AAA几何结构。通常,每个患者的4D-CTA数据集包含在整个心脏周期内获取的心电图门控的3D-CTA图像帧,这些图像捕捉AAA配置的收缩期和舒张期。为了验证方法,该数据集还包括由患者1的舒张期3D-CTA AAA图像生成的合成基准数据。基准数据包括患者特定的有限元(FE)生物力学模型和合成的收缩期3D-CTA图像。合成收缩期图像是通过使用来自AAA生物力学有限元模型的现实位移场对患者1的舒张期3D-CTA图像进行变形处理而生成的。这些图像是在西澳大利亚州的菲欧娜斯坦利医院获取的,并提供给西澳大利亚大学智能医学实验室(ISML-UWA)的研究人员,在那里进行了基于图像的AAA运动学分析。我们的数据集能够使用一种无创、体内、基于图像配准的方法分析整个心脏周期内AAA壁位移和应变。使用广泛采用的开放源代码文件格式(NRRD用于图像和STL用于几何形状)有助于在AAA生物力学研究中实现广泛的适用性和可重用性,这些研究需要特定于患者的几何形状以及有关心脏周期内AAA运动学的信息。
论文及项目相关链接
Summary
该文章介绍了一个用于研究腹部主动脉瘤(AAA)运动学的数据集。数据集可从Zenodo数据仓库下载,包含通过4D-CTA技术获取的AAA患者的实时三维图像以及从这些图像中提取的患者特异性AAA几何结构。数据集还包括合成基准数据,用于验证方法。该数据集有助于分析AAA壁在整个心动周期中的位移和应变,采用非侵入性、活体、基于图像配准的方法。数据集使用广泛采纳的开放源文件格式,便于在AAA生物力学研究中应用。
Key Takeaways
- 文章介绍了一个关于腹部主动脉瘤(AAA)运动学研究的公开数据集。
- 数据集包含通过4D-CTA技术获取的AAA患者的实时三维图像。
- 数据集包含患者特异性AAA几何结构以及合成基准数据用于方法验证。
- 数据集可用于分析AAA壁在整个心动周期中的位移和应变。
- 数据集采用了非侵入性、活体、基于图像配准的方法进行分析。
- 数据集使用开放源文件格式,便于在AAA生物力学研究中使用。
点此查看论文截图

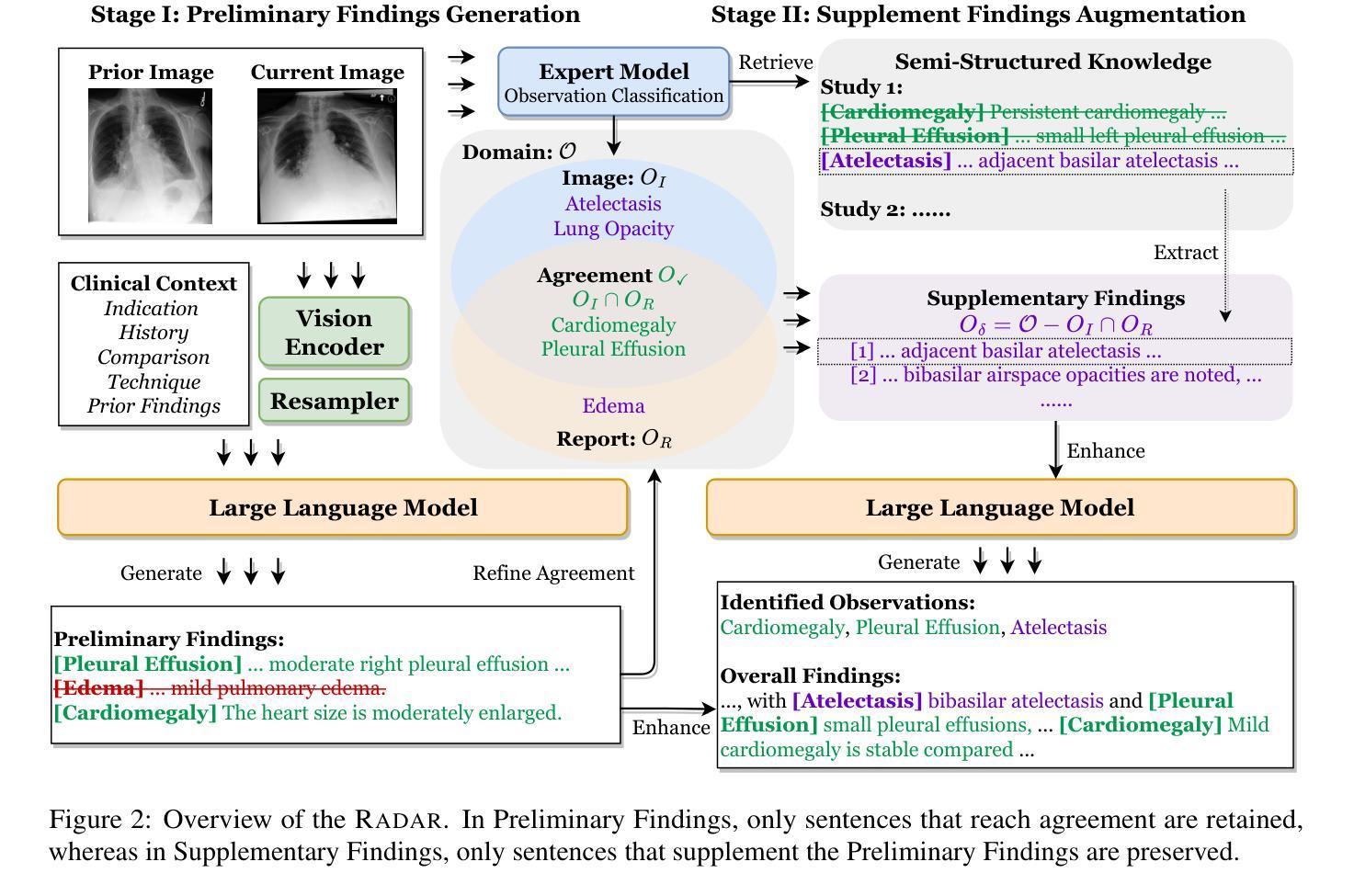
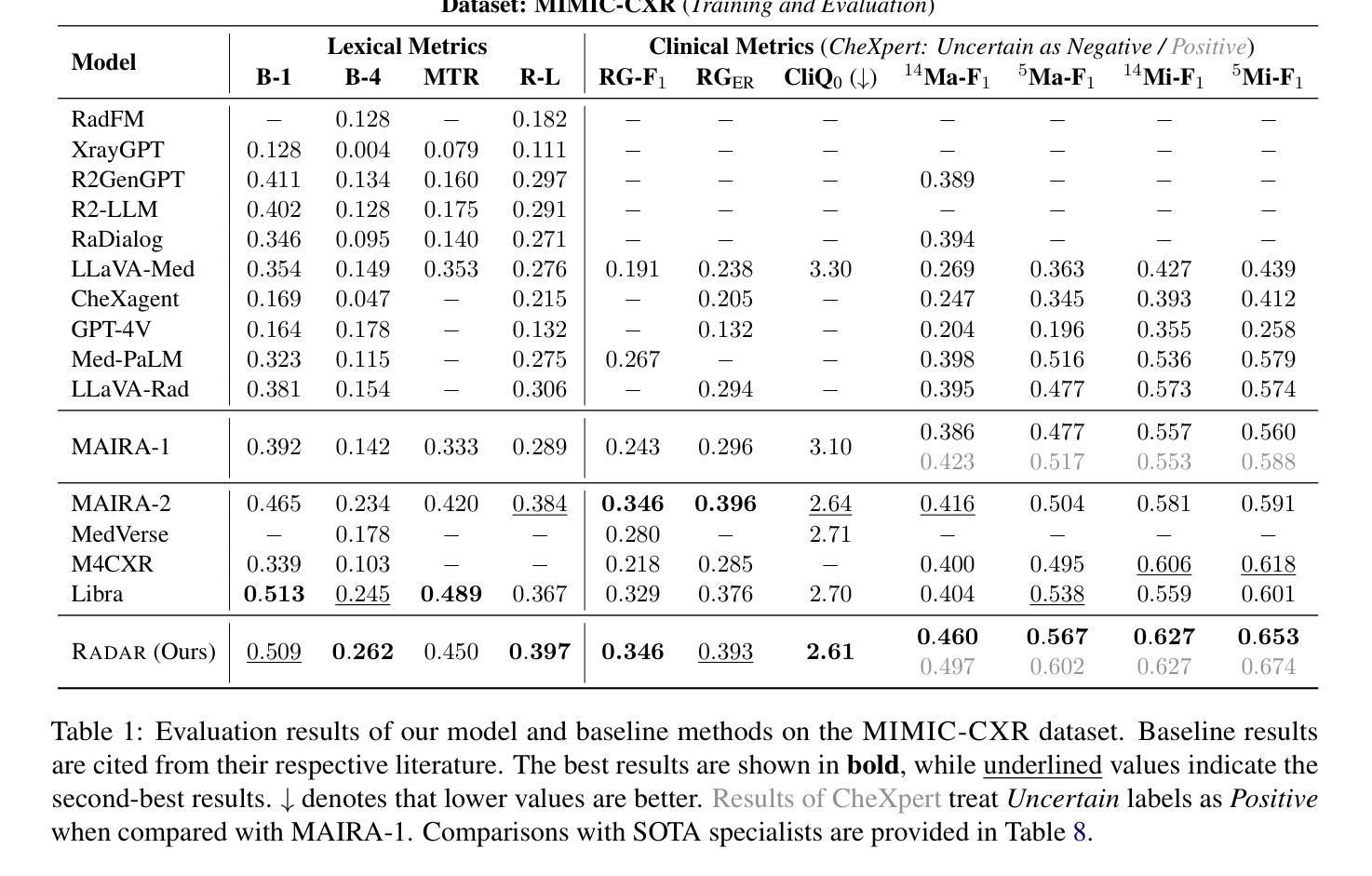
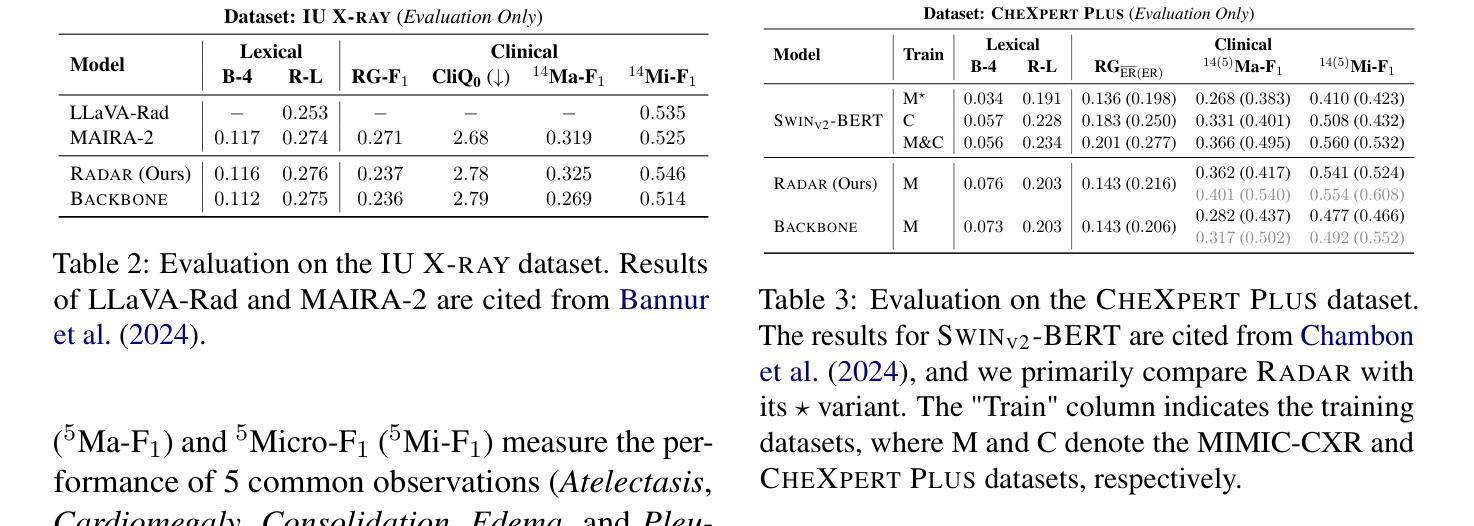
RADAR: Enhancing Radiology Report Generation with Supplementary Knowledge Injection
Authors:Wenjun Hou, Yi Cheng, Kaishuai Xu, Heng Li, Yan Hu, Wenjie Li, Jiang Liu
Large language models (LLMs) have demonstrated remarkable capabilities in various domains, including radiology report generation. Previous approaches have attempted to utilize multimodal LLMs for this task, enhancing their performance through the integration of domain-specific knowledge retrieval. However, these approaches often overlook the knowledge already embedded within the LLMs, leading to redundant information integration. To address this limitation, we propose Radar, a framework for enhancing radiology report generation with supplementary knowledge injection. Radar improves report generation by systematically leveraging both the internal knowledge of an LLM and externally retrieved information. Specifically, it first extracts the model’s acquired knowledge that aligns with expert image-based classification outputs. It then retrieves relevant supplementary knowledge to further enrich this information. Finally, by aggregating both sources, Radar generates more accurate and informative radiology reports. Extensive experiments on MIMIC-CXR, CheXpert-Plus, and IU X-ray demonstrate that our model outperforms state-of-the-art LLMs in both language quality and clinical accuracy.
大型语言模型(LLM)在多个领域表现出了卓越的能力,包括放射学报告生成。之前的方法试图使用多模态LLM来完成此任务,通过整合特定领域的知识检索来增强性能。然而,这些方法往往会忽略LLM中已经嵌入的知识,导致信息集成冗余。为了解决这一局限性,我们提出了Radar框架,通过注入补充知识来提高放射学报告的生成质量。Radar通过系统地利用LLM的内部知识和外部检索信息来改善报告生成。具体来说,它首先提取与专家基于图像的分类输出相符的模型获取的知识。然后,它检索相关的补充知识来进一步丰富这些信息。最后,通过聚合这两个来源,Radar生成更准确和更有信息的放射学报告。在MIMIC-CXR、CheXpert-Plus和IU X-ray上的广泛实验表明,我们的模型在语言质量和临床准确性方面都优于最新的LLM。
论文及项目相关链接
PDF Accepted to ACL 2025 main
摘要
大规模语言模型(LLMs)在多个领域表现出卓越的能力,包括放射学报告生成。先前的方法试图使用多模式LLMs完成此任务,并通过整合领域特定知识检索增强其性能。然而,这些方法往往忽视了LLMs中已经嵌入的知识,导致信息整合冗余。为解决这一局限性,我们提出了Radar框架,用于通过补充知识注入增强放射学报告生成。Radar通过系统地利用LLM的内部知识和外部检索信息,改进了报告生成。具体而言,它首先提取与专家图像分类输出对齐的模型获取的知识。然后,它检索相关的补充知识以进一步丰富这些信息。最后,通过聚合这两个来源,Radar生成更准确和全面的放射学报告。在MIMIC-CXR、CheXpert-Plus和IU X-ray上的广泛实验表明,我们的模型在语言质量和临床准确性方面均优于最新LLMs。
关键见解
- 大规模语言模型在放射学报告生成等领域具有显著能力。
- 先前的方法通过整合领域特定知识检索来增强性能,但忽视了LLMs中已嵌入的知识。
- Radar框架通过系统地利用LLM的内部知识和外部检索信息,改进了放射学报告生成。
- Radar提取与专家图像分类输出对齐的模型获取的知识,并检索相关补充知识。
- 通过聚合模型内部知识和外部检索信息,Radar能生成更准确和全面的放射学报告。
- 广泛实验证明,Radar在语言和临床准确性方面优于其他最新LLMs。
- Radar框架有助于实现更高效的放射学报告生成,具有潜在的临床应用价值。
点此查看论文截图

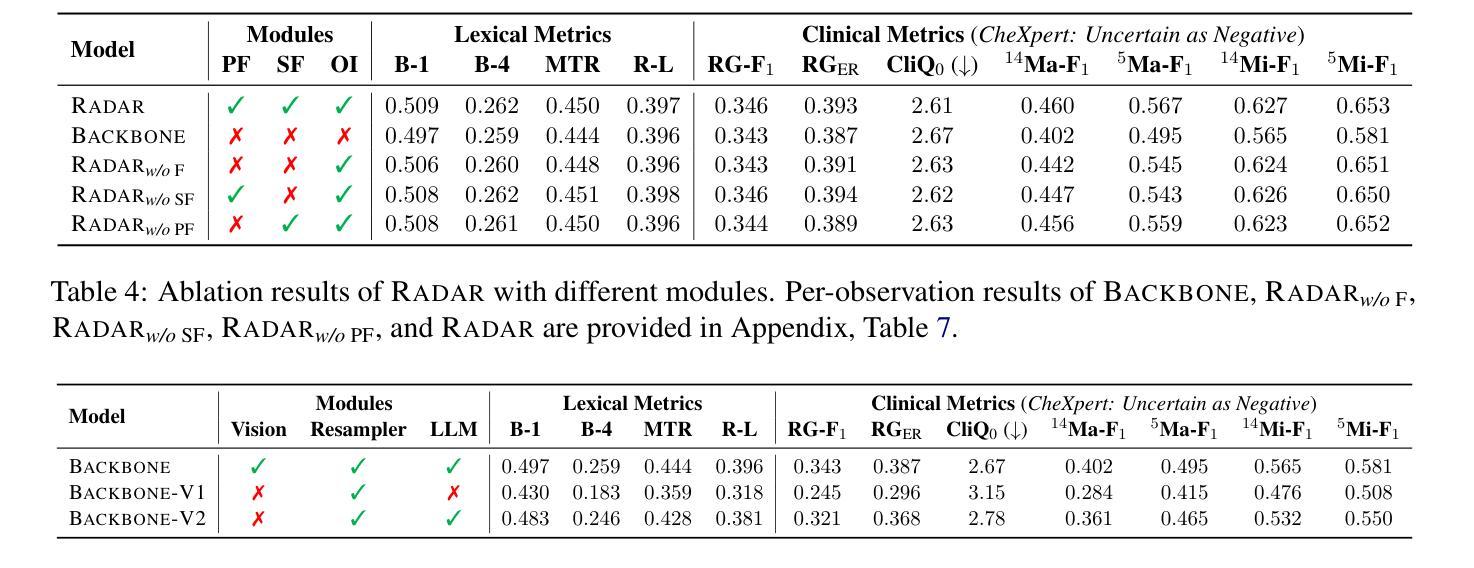
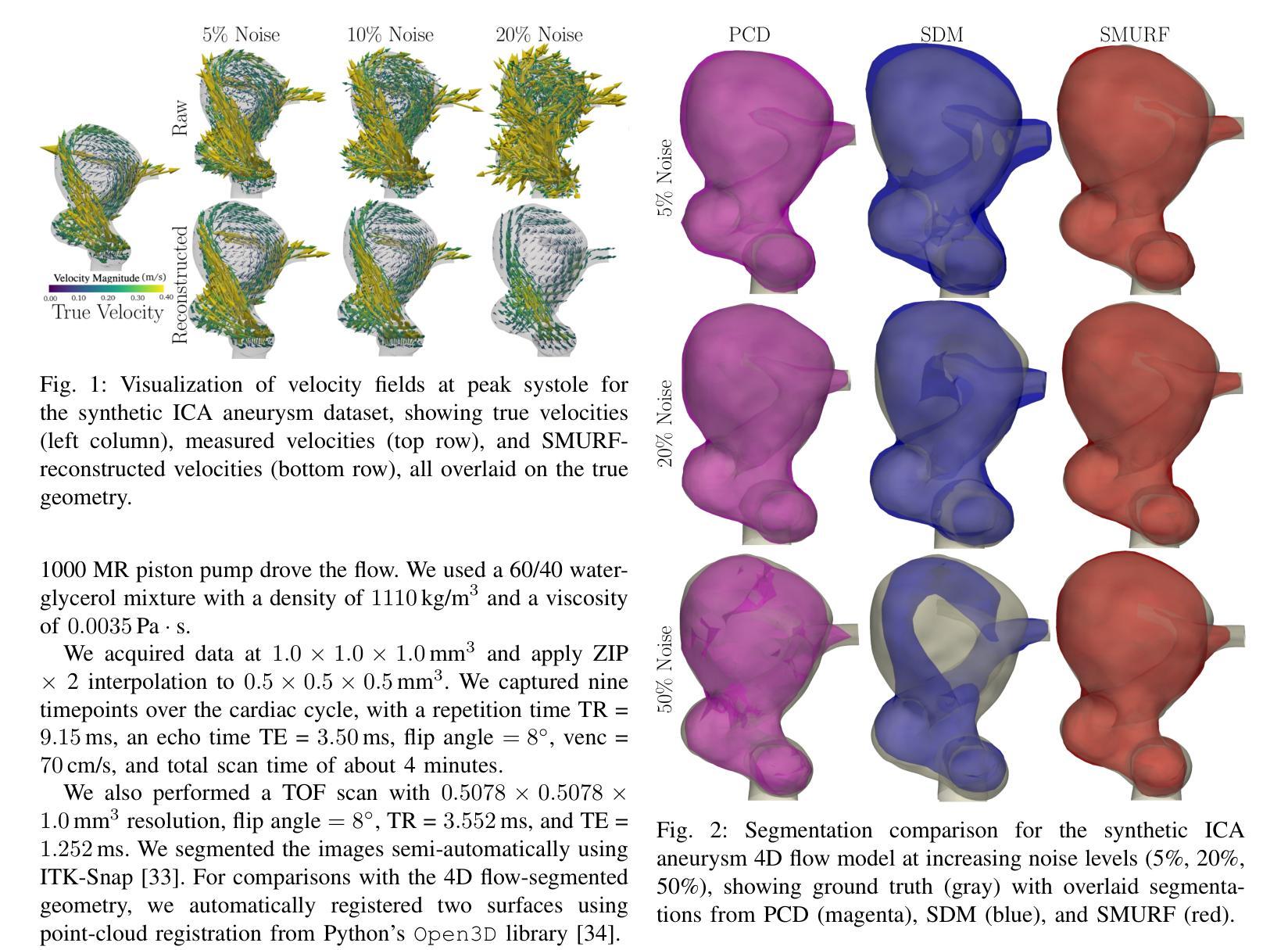
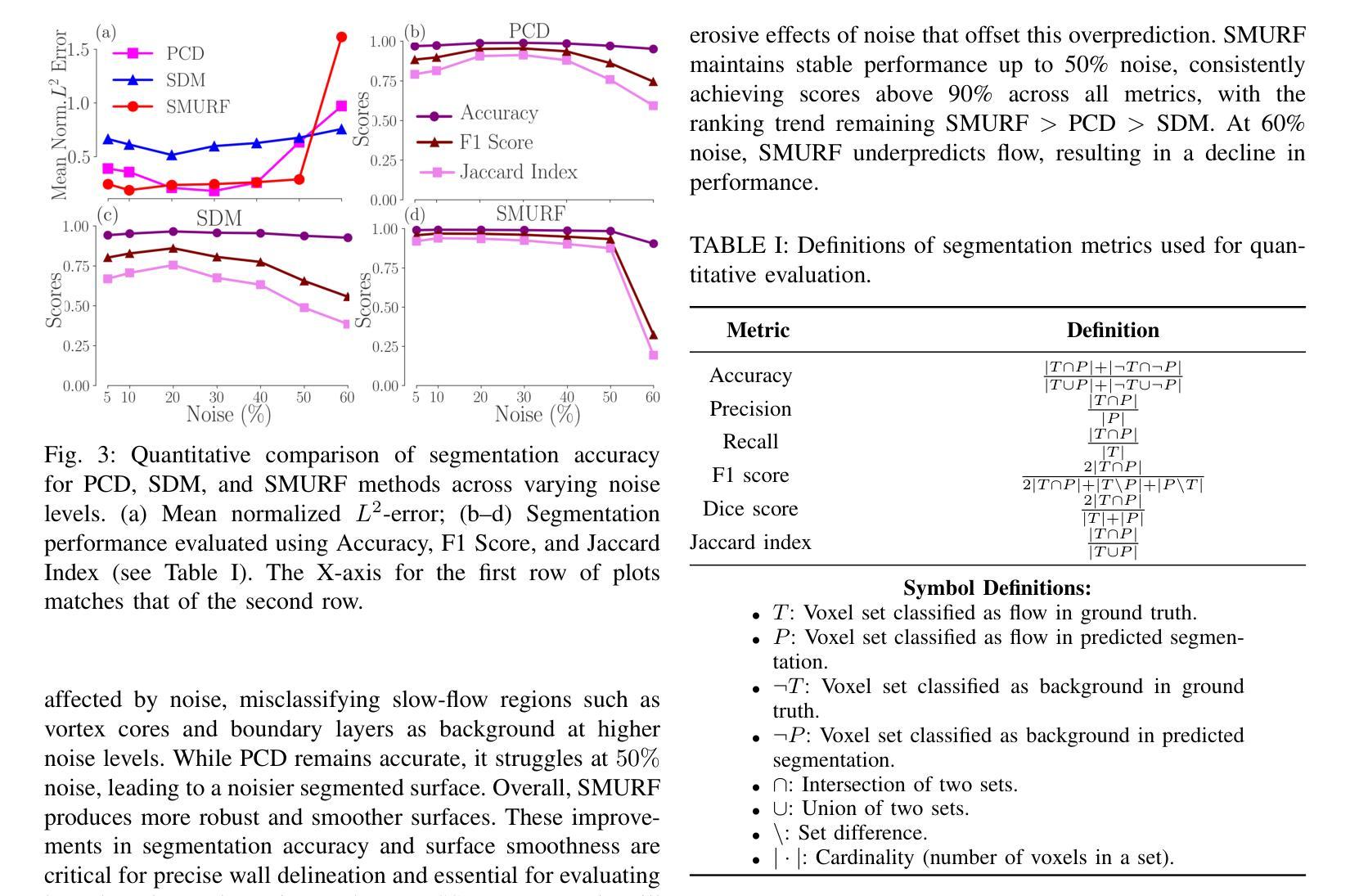
SMURF: Scalable method for unsupervised reconstruction of flow in 4D flow MRI
Authors:Atharva Hans, Abhishek Singh, Pavlos Vlachos, Ilias Bilionis
We introduce SMURF, a scalable and unsupervised machine learning method for simultaneously segmenting vascular geometries and reconstructing velocity fields from 4D flow MRI data. SMURF models geometry and velocity fields using multilayer perceptron-based functions incorporating Fourier feature embeddings and random weight factorization to accelerate convergence. A measurement model connects these fields to the observed image magnitude and phase data. Maximum likelihood estimation and subsampling enable SMURF to process high-dimensional datasets efficiently. Evaluations on synthetic, in vitro, and in vivo datasets demonstrate SMURF’s performance. On synthetic internal carotid artery aneurysm data derived from CFD, SMURF achieves a quarter-voxel segmentation accuracy across noise levels of up to 50%, outperforming the state-of-the-art segmentation method by up to double the accuracy. In an in vitro experiment on Poiseuille flow, SMURF reduces velocity reconstruction RMSE by approximately 34% compared to raw measurements. In in vivo internal carotid artery aneurysm data, SMURF attains nearly half-voxel segmentation accuracy relative to expert annotations and decreases median velocity divergence residuals by about 31%, with a 27% reduction in the interquartile range. These results indicate that SMURF is robust to noise, preserves flow structure, and identifies patient-specific morphological features. SMURF advances 4D flow MRI accuracy, potentially enhancing the diagnostic utility of 4D flow MRI in clinical applications.
我们介绍SMURF,这是一种可扩展的无监督机器学习方法,能够同时从4D流MRI数据中分割血管几何结构并重建速度场。SMURF使用多层感知器函数对几何形状和速度场进行建模,并结合傅里叶特征嵌入和随机权重分解来加速收敛。测量模型将这些场与观察到的图像幅度和相位数据连接起来。最大似然估计和子采样使SMURF能够高效处理高维数据集。在合成、体外和体内数据集上的评估证明了SMURF的性能。在由CFD派生的合成内部颈动脉动脉瘤数据上,SMURF在高达50%的噪声水平下实现了四分之一体素的分割精度,比最新分割方法的精度高出两倍。在泊松流的体外实验中,SMURF与原始测量相比,速度重建的RMSE降低了约34%。在体内的内部颈动脉动脉瘤数据中,SMURF相对于专家注释达到了近一半体素的分割精度,并将中位数速度发散残差减少了约31%,四分位距减少了27%。这些结果表明,SMURF对噪声具有鲁棒性,能够保留流动结构,并识别患者特定的形态特征。SMURF提高了4D流MRI的准确性,有望增强4D流MRI在临床应用中的诊断效用。
论文及项目相关链接
Summary
SMURF是一种可扩展的无监督机器学习方法,可同时实现血管几何结构的分割和速度场的重建,适用于处理四维血流MRI数据。SMURF采用多层感知器函数建模几何和速度场,并结合傅里叶特征嵌入和随机权重分解加速收敛。其在合成、体外和体内数据集上的评估证明了其性能优势。SMURF具有抗噪性强、保留血流结构以及识别患者特定形态特征等优点,提高了四维血流MRI的准确性,有望增强其在临床应用中的诊断价值。
Key Takeaways
- SMURF是一种用于处理四维血流MRI数据的可扩展无监督机器学习技术。
- SMURF能同时分割血管几何结构和重建速度场。
- SMURF采用多层感知器结合傅里叶特征嵌入和随机权重分解来建模。
- 在合成数据集上,SMURF在噪声水平高达50%的情况下实现了四分之一体素的分割精度,优于现有技术。
- 在体外实验中,SMURF将速度重建的RMSE降低了约34%。
- 在体内数据集上,SMURF达到了近乎一半体素的分割精度,并降低了速度散度残差。
点此查看论文截图

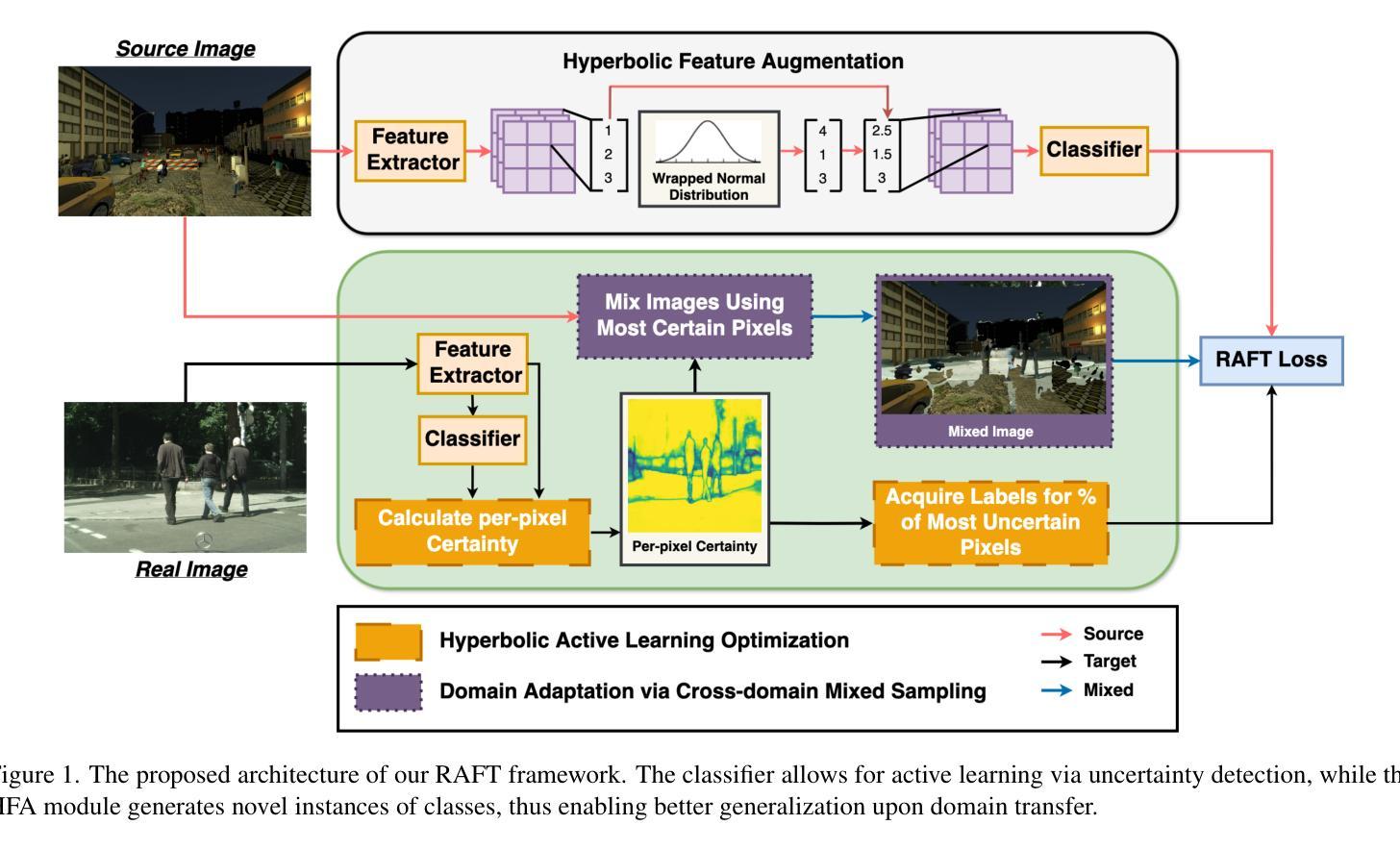
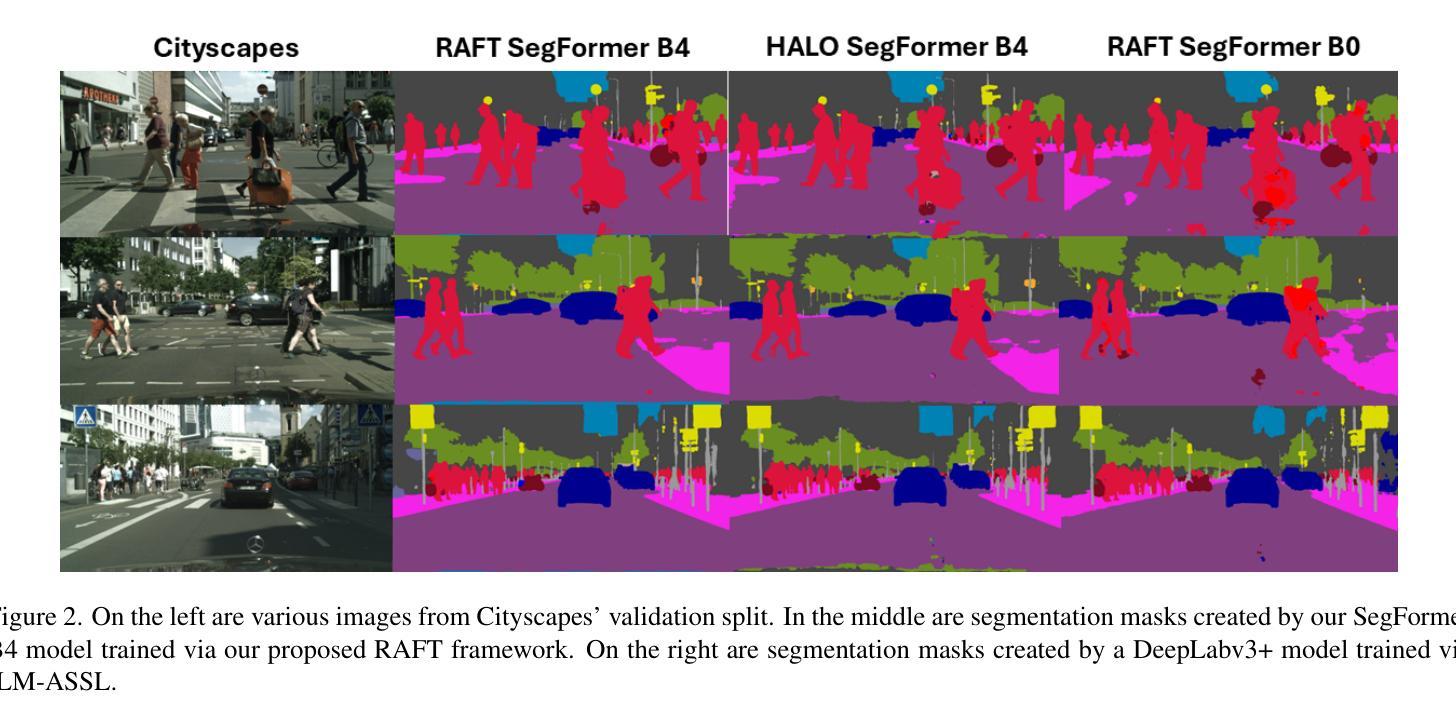
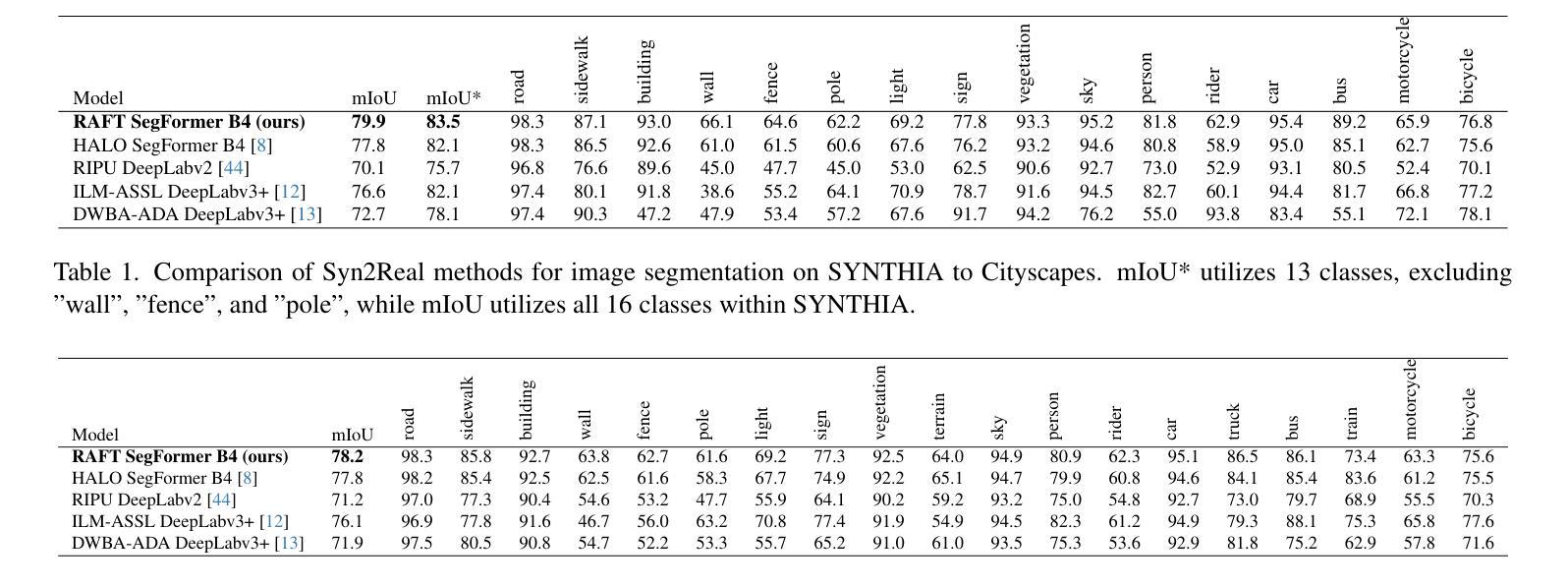
RAFT: Robust Augmentation of FeaTures for Image Segmentation
Authors:Edward Humes, Xiaomin Lin, Uttej Kallakuri, Tinoosh Mohsenin
Image segmentation is a powerful computer vision technique for scene understanding. However, real-world deployment is stymied by the need for high-quality, meticulously labeled datasets. Synthetic data provides high-quality labels while reducing the need for manual data collection and annotation. However, deep neural networks trained on synthetic data often face the Syn2Real problem, leading to poor performance in real-world deployments. To mitigate the aforementioned gap in image segmentation, we propose RAFT, a novel framework for adapting image segmentation models using minimal labeled real-world data through data and feature augmentations, as well as active learning. To validate RAFT, we perform experiments on the synthetic-to-real “SYNTHIA->Cityscapes” and “GTAV->Cityscapes” benchmarks. We managed to surpass the previous state of the art, HALO. SYNTHIA->Cityscapes experiences an improvement in mIoU* upon domain adaptation of 2.1%/79.9%, and GTAV->Cityscapes experiences a 0.4%/78.2% improvement in mIoU. Furthermore, we test our approach on the real-to-real benchmark of “Cityscapes->ACDC”, and again surpass HALO, with a gain in mIoU upon adaptation of 1.3%/73.2%. Finally, we examine the effect of the allocated annotation budget and various components of RAFT upon the final transfer mIoU.
图像分割是场景理解的一种强大的计算机视觉技术。然而,实际部署却受到需要高质量、精细标注数据集的限制。合成数据提供了高质量标签,同时减少了手动数据收集和标注的需求。然而,在合成数据上训练的深度神经网络常常面临Syn2Real问题,导致在实际部署中的性能不佳。为了减轻上述图像分割中的差距,我们提出了RAFT,这是一个使用最小量的真实世界数据,通过数据和特征增强以及主动学习,适应图像分割模型的新框架。为了验证RAFT的效果,我们在合成到真实的”SYNTHIA->Cityscapes”和”GTAV->Cityscapes”基准测试集上进行了实验。我们超越了之前的技术水平HALO。在SYNTHIA->Cityscapes上,我们的域适应方法提高了mIoU*(平均交并比)的准确度达到2.1%/79.9%,而在GTAV->Cityscapes上提高了mIoU的准确度达到0.4%/78.2%。此外,我们在真实到真实的”Cityscapes->ACDC”基准测试集上测试了我们的方法,并再次超越了HALO,在适应后提高了mIoU的准确度达到1.3%/73.2%。最后,我们研究了分配标注预算对RAFT最终迁移mIoU的影响以及各种组件的影响。
论文及项目相关链接
Summary
图像分割是一种强大的计算机视觉场景理解技术,但在实际部署中需要高质量、精细标注的数据集。合成数据可以提供高质量标签,减少手动收集和标注的需求。然而,在合成数据上训练的深度神经网络常面临合成到真实(Syn2Real)问题,导致在真实世界部署中的性能不佳。为缓解图像分割中的前述差距,我们提出RAFT框架,通过最小真实世界数据、数据增强和特征增强以及主动学习方法,适应图像分割模型。验证实验表明,RAFT在合成到真实场景的基准测试中表现超越先前技术。
Key Takeaways
- 图像分割是计算机视觉中的一项重要技术,用于场景理解。
- 实际部署图像分割技术时,需要高质量、精细标注的数据集。
- 合成数据可以减少手动收集和标注数据的需求,但训练的模型在真实世界部署中常面临性能问题。
- 提出的RAFT框架旨在通过最小真实世界数据、数据增强和特征增强以及主动学习方法,适应图像分割模型。
- RAFT在合成到真实场景的基准测试中表现超越先前技术。
- 在SYNTHIA->Cityscapes和GTAV->Cityscapes的基准测试中,RAFT在mIoU指标上实现了显著的改进。
点此查看论文截图

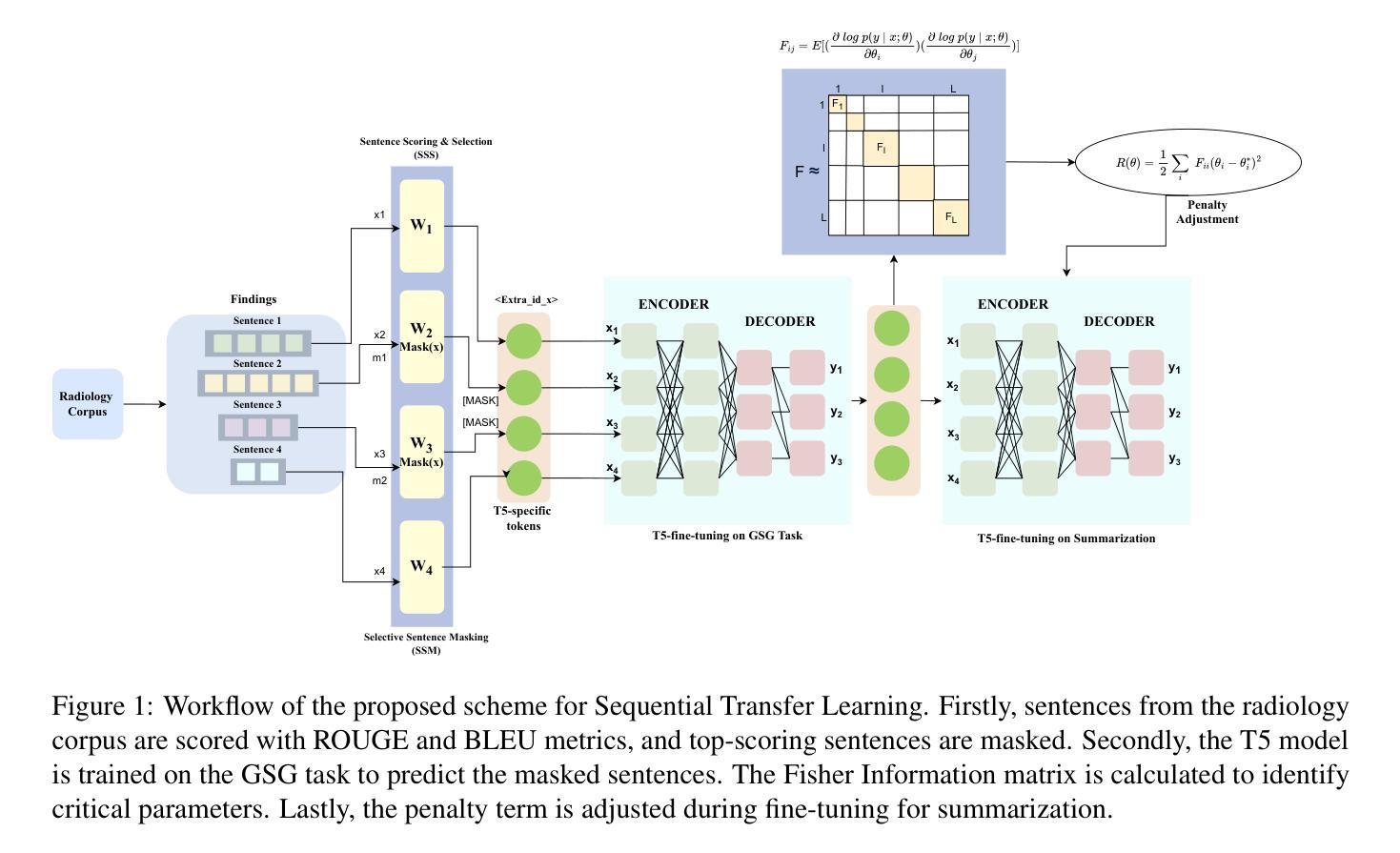
CSTRL: Context-Driven Sequential Transfer Learning for Abstractive Radiology Report Summarization
Authors:Mst. Fahmida Sultana Naznin, Adnan Ibney Faruq, Mostafa Rifat Tazwar, Md Jobayer, Md. Mehedi Hasan Shawon, Md Rakibul Hasan
A radiology report comprises several sections, including the Findings and Impression of the diagnosis. Automatically generating the Impression from the Findings is crucial for reducing radiologists’ workload and improving diagnostic accuracy. Pretrained models that excel in common abstractive summarization problems encounter challenges when applied to specialized medical domains largely due to the complex terminology and the necessity for accurate clinical context. Such tasks in medical domains demand extracting core information, avoiding context shifts, and maintaining proper flow. Misuse of medical terms can lead to drastic clinical errors. To address these issues, we introduce a sequential transfer learning that ensures key content extraction and coherent summarization. Sequential transfer learning often faces challenges like initial parameter decay and knowledge loss, which we resolve with the Fisher matrix regularization. Using MIMIC-CXR and Open-I datasets, our model, CSTRL - Context-driven Sequential TRansfer Learning - achieved state-of-the-art performance, showing 56.2% improvement in BLEU-1, 40.5% in BLEU-2, 84.3% in BLEU-3, 28.9% in ROUGE-1, 41.0% in ROUGE-2 and 26.5% in ROGUE-3 score over benchmark studies. We also analyze factual consistency scores while preserving the medical context. Our code is publicly available at https://github.com/fahmidahossain/Report_Summarization.
医学放射报告由几个部分组成,包括诊断的检查结果和印象部分。自动根据检查结果生成诊断印象对减少放射科医生的工作量、提高诊断准确性至关重要。预训练模型在常见的抽象摘要问题中表现优异,但当应用于专业医学领域时,由于复杂的术语和准确的临床语境的必要性,它们面临挑战。此类医学领域的任务需要提取核心信息,避免上下文偏移,并保持适当的流程。医学术语的误用可能导致严重的临床错误。为了解决这些问题,我们引入了一种顺序迁移学习,以确保关键内容提取和连贯的摘要。顺序迁移学习经常面临初始参数衰减和知识损失等挑战,我们通过Fisher矩阵正则化来解决这些问题。使用MIMIC-CXR和Open-I数据集,我们的模型CSTRL(基于上下文驱动的序列迁移学习)取得了最先进的性能表现。相较于基准研究,BLEU-1得分提高了56.2%,BLEU-2得分提高了40.5%,BLEU-3得分提高了84.3%,在ROUGE得分方面也有所提高。我们还分析了保持医学上下文的一致度得分。我们的代码公开在https://github.com/fahmidahossain/Report_Summarization。
论文及项目相关链接
PDF Accepted in ACL 2025 Findings
Summary
本文主要介绍了一种针对医学图像报告自动摘要的技术。通过使用序贯迁移学习,该技术在医学领域的文本摘要中实现了显著提升,可以有效减少医生的工作量并提升诊断的准确性。模型的摘要结果公开可访问。文中提到了挑战与解决策略以及模型效果评估结果。
Key Takeaways
- 文章介绍了一种基于序贯迁移学习的医学图像报告自动摘要技术。
- 该技术解决了传统模型在医学领域文本摘要中面临的挑战,如术语复杂度和准确临床语境的把握。
- 该方法能够在关键内容提取和连贯性总结之间实现平衡,避免因医学术语误用导致的临床错误。
- 通过使用Fisher矩阵正则化,解决了序贯迁移学习中初始参数衰减和知识损失的问题。
- 模型在MIMIC-CXR和Open-I数据集上进行了实验验证,取得了显著的提升效果。相较于基准研究,模型在BLEU和ROUGE得分上有显著提高。
- 文章对模型的性能进行了详细评估,包括事实一致性评估,确保在保留医学语境的同时提高摘要的准确性。
点此查看论文截图

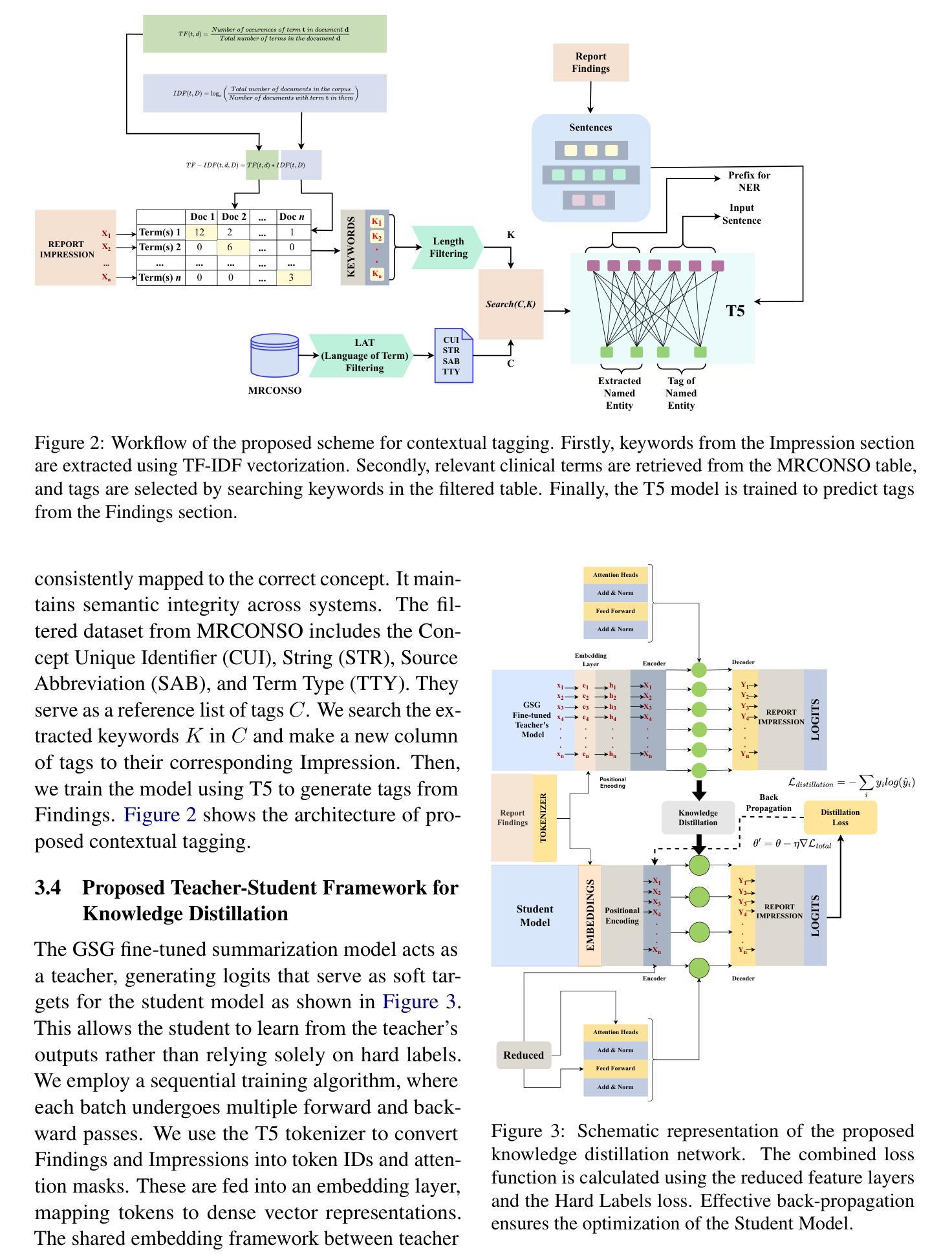
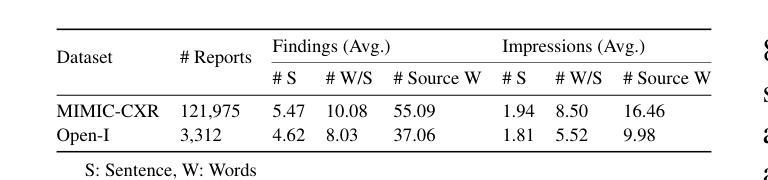
Fourier Asymmetric Attention on Domain Generalization for Pan-Cancer Drug Response Prediction
Authors:Ran Song, Yinpu Bai, Hui Liu
The accurate prediction of drug responses remains a formidable challenge, particularly at the single-cell level and in clinical treatment contexts. Some studies employ transfer learning techniques to predict drug responses in individual cells and patients, but they require access to target-domain data during training, which is often unavailable or only obtainable in future. In this study, we propose a novel domain generalization framework, termed FourierDrug, to address this challenge. Given the extracted feature from expression profile, we performed Fourier transforms and then introduced an asymmetric attention constraint that would cluster drug-sensitive samples into a compact group while drives resistant samples dispersed in the frequency domain. Our empirical experiments demonstrate that our model effectively learns task-relevant features from diverse source domains, and achieves accurate predictions of drug response for unseen cancer type. When evaluated on single-cell and patient-level drug response prediction tasks, FourierDrug–trained solely on in vitro cell line data without access to target-domain data–consistently outperforms or, at least, matched the performance of current state-of-the-art methods. These findings underscore the potential of our method for real-world clinical applications.
准确预测药物反应仍然是一项艰巨的挑战,特别是在单细胞水平和临床治疗环境中。一些研究采用迁移学习技术来预测单个细胞和患者的药物反应,但它们需要在训练过程中访问目标域数据,而这些数据通常不可用或只能在将来获得。本研究提出了一种新的领域泛化框架,称为FourierDrug,以解决这一挑战。根据表达谱提取的特征,我们进行了傅里叶变换,然后引入了一个不对称注意力约束,该约束会将药物敏感样本聚集成一个紧凑的群体,同时使耐药样本在频域中分散。我们的经验实验表明,我们的模型有效地从多个源域学习了任务相关特征,并对未见过的癌症类型实现了准确的药物反应预测。在单细胞和患者水平的药物反应预测任务上评估时,FourierDrug(仅在体外细胞系数据上训练,无需访问目标域数据)始终优于或至少与当前最新方法的表现相匹配。这些发现突出了我们的方法在实际临床应用中的潜力。
论文及项目相关链接
Summary
医学领域预测药物反应仍是重大挑战,特别是在单细胞层面和临床治疗环境中。本研究提出了一种名为FourierDrug的新型域泛化框架,通过傅里叶变换和不对称注意力约束,能在未接触目标域数据的情况下,有效学习来自不同源域的任务相关特征,并对未见过的癌症类型进行药物反应预测。在单细胞和患者层面的药物反应预测任务中,FourierDrug的表现优于或至少与当前最先进的方法相匹配。
Key Takeaways
- 医学领域预测药物反应是一大挑战,特别是在单细胞和临床治疗环境中。
- 研究提出了名为FourierDrug的新型域泛化框架来解决此挑战。
- FourierDrug利用傅里叶变换和不对称注意力约束进行特征提取和分类。
- 该模型可在未接触目标域数据的情况下,学习来自不同源域的任务相关特征。
- FourierDrug在未见过的癌症类型药物反应预测中表现出优异性能。
- 在单细胞和患者层面的药物反应预测任务中,FourierDrug的表现优于或至少与当前最先进的方法相匹配。
点此查看论文截图

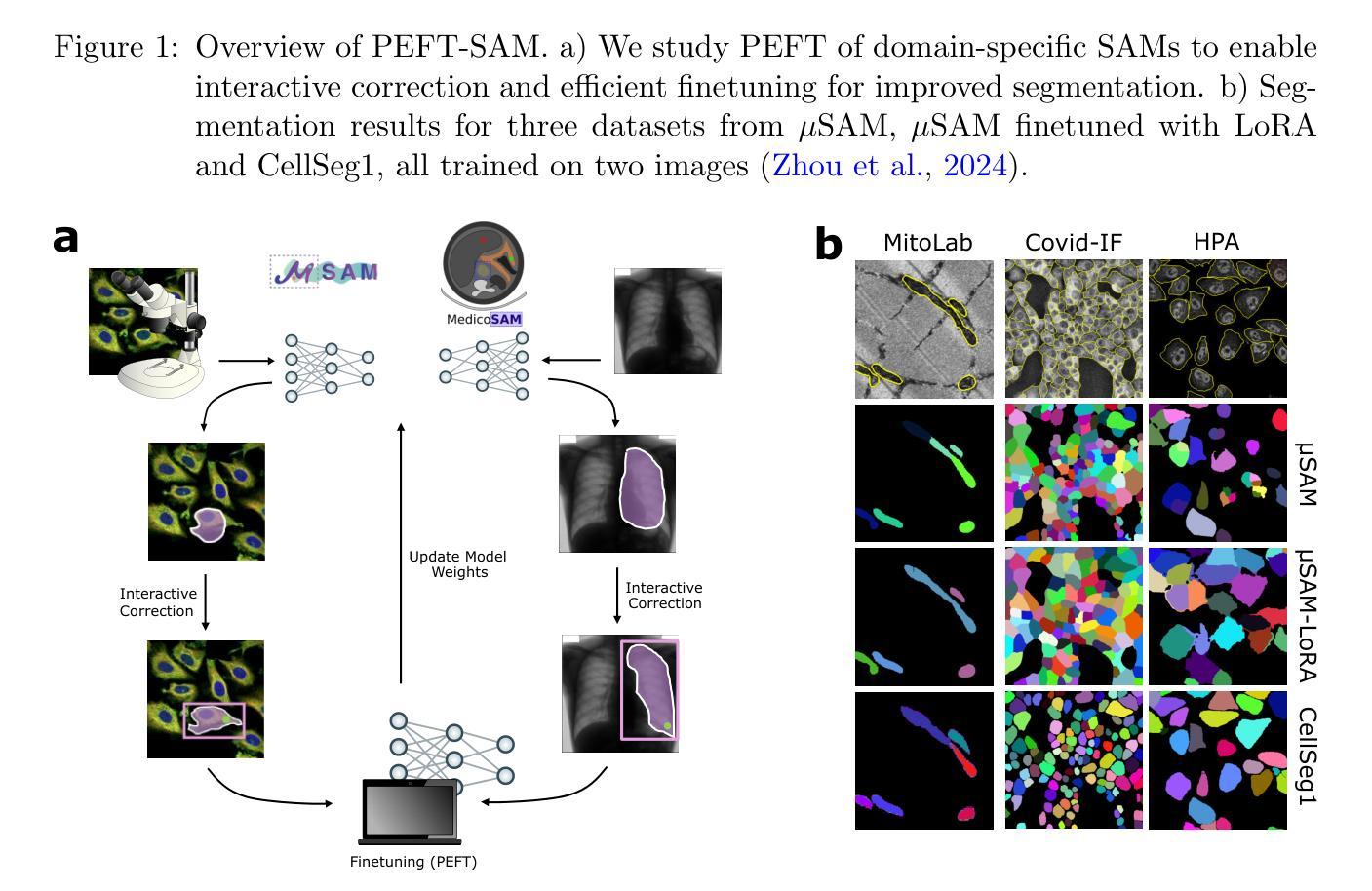
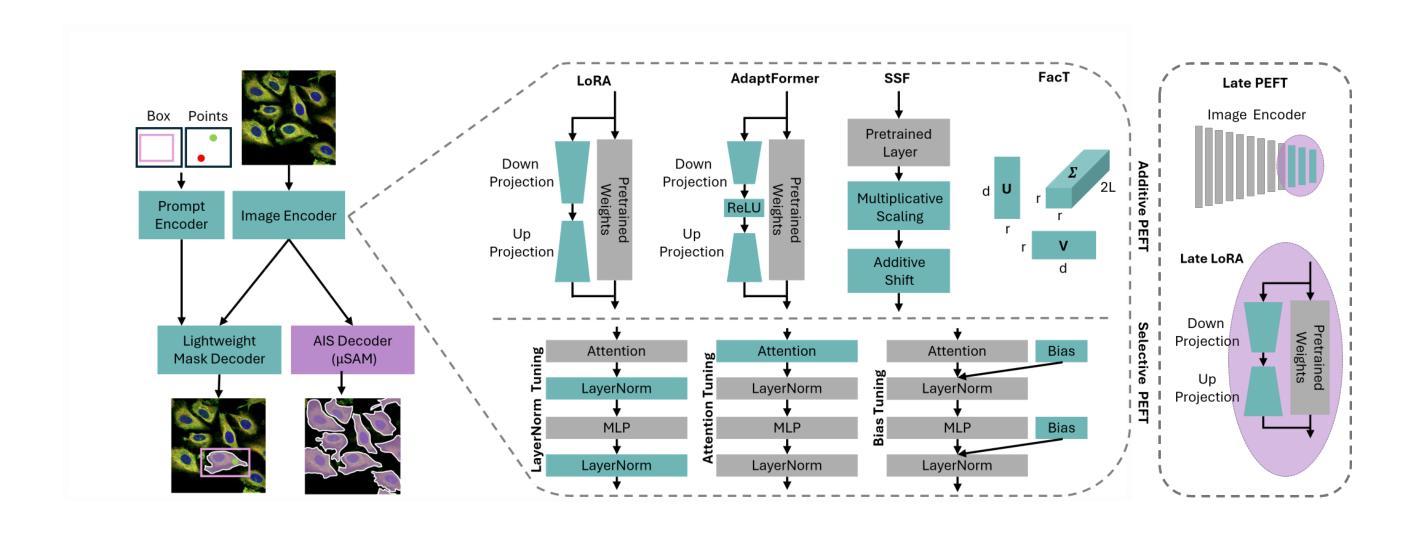
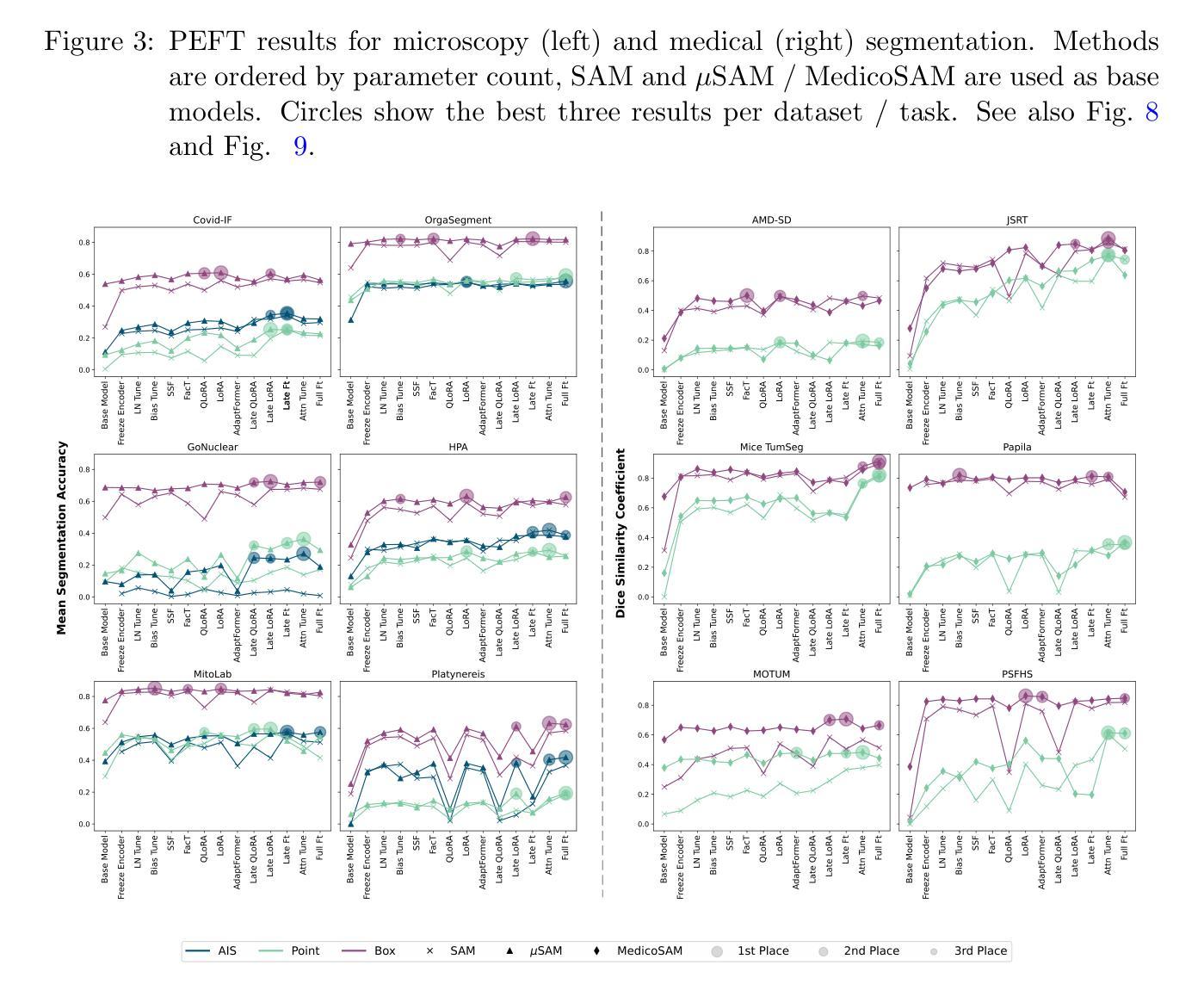
Parameter Efficient Fine-Tuning of Segment Anything Model for Biomedical Imaging
Authors:Carolin Teuber, Anwai Archit, Constantin Pape
Segmentation is an important analysis task for biomedical images, enabling the study of individual organelles, cells or organs. Deep learning has massively improved segmentation methods, but challenges remain in generalization to new conditions, requiring costly data annotation. Vision foundation models, such as Segment Anything Model (SAM), address this issue through improved generalization. However, these models still require finetuning on annotated data, although with less annotations, to achieve optimal results for new conditions. As a downside, they require more computational resources. This makes parameter-efficient finetuning (PEFT) relevant. We contribute the first comprehensive study of PEFT for SAM applied to biomedical images. We find that the placement of PEFT layers is more important for efficiency than the type of layer for vision transformers and we provide a recipe for resource-efficient finetuning. Our code is publicly available at https://github.com/computational-cell-analytics/peft-sam.
分割是生物医学图像分析的重要任务,能够对单个细胞器、细胞或器官进行研究。深度学习极大地改善了分割方法,但仍然存在对新条件泛化方面的挑战,这需要昂贵的标注数据。视觉基础模型(如任何分割模型(SAM))通过提高泛化能力来解决这个问题。然而,这些模型虽然使用较少的标注数据,但仍需在标注数据上进行微调,以实现新条件下的最佳结果。作为代价,它们需要更多的计算资源。这使参数高效微调(PEFT)变得至关重要。我们对应用于生物医学图像的SAM的PEFT进行了首次全面研究。我们发现对于视觉转换器而言,PEFT层的放置比层的类型对效率更重要,并为资源高效的微调提供了一个配方。我们的代码公开在https://github.com/computational-cell-analytics/peft-sam。
论文及项目相关链接
PDF Published in MIDL 2025
Summary
深度学习在生物医学图像分割方法中发挥了巨大作用,但仍存在对新条件泛化能力有限的问题,需要昂贵的数据标注成本。Segment Anything Model(SAM)等视觉基础模型通过改进泛化能力来解决这一问题,但仍需在标注数据上进行微调以达到最佳效果。参数高效微调(PEFT)是一种解决该问题的方法,本研究首次全面研究了PEFT在生物医学图像中应用于SAM的效果,发现对于视觉转换器而言,PEFT层的放置比层的类型更重要,并提供了一种资源高效的微调配方。相关代码已公开于https://github.com/computational-cell-analytics/peft-sam。
Key Takeaways
- 深度学习在生物医学图像分割中发挥着重要作用,但仍面临对新条件泛化能力和数据标注成本的问题。
- Segment Anything Model(SAM)通过改进泛化能力来解决这些问题,但仍需微调以达到最佳效果。
- 参数高效微调(PEFT)是一种解决微调所需大量计算资源的方法。
- PEFT在生物医学图像中应用于SAM的效果研究指出,PEFT层的放置比层的类型更重要。
- 研究提供了一种资源高效的微调配方。
- 该研究的相关代码已公开,便于其他研究者使用。
点此查看论文截图

Segment Anything for Histopathology
Authors:Titus Griebel, Anwai Archit, Constantin Pape
Nucleus segmentation is an important analysis task in digital pathology. However, methods for automatic segmentation often struggle with new data from a different distribution, requiring users to manually annotate nuclei and retrain data-specific models. Vision foundation models (VFMs), such as the Segment Anything Model (SAM), offer a more robust alternative for automatic and interactive segmentation. Despite their success in natural images, a foundation model for nucleus segmentation in histopathology is still missing. Initial efforts to adapt SAM have shown some success, but did not yet introduce a comprehensive model for diverse segmentation tasks. To close this gap, we introduce PathoSAM, a VFM for nucleus segmentation, based on training SAM on a diverse dataset. Our extensive experiments show that it is the new state-of-the-art model for automatic and interactive nucleus instance segmentation in histopathology. We also demonstrate how it can be adapted for other segmentation tasks, including semantic nucleus segmentation. For this task, we show that it yields results better than popular methods, while not yet beating the state-of-the-art, CellViT. Our models are open-source and compatible with popular tools for data annotation. We also provide scripts for whole-slide image segmentation. Our code and models are publicly available at https://github.com/computational-cell-analytics/patho-sam.
细胞核分割是数字病理学中的重要分析任务。然而,自动分割方法通常难以处理来自不同分布的新数据,需要用户手动注释细胞核并重新训练特定数据模型。例如分段任何模型(SAM)的视野基础模型(VFMs)为自动和交互式分割提供了更稳健的替代方案。尽管它们在自然图像中取得了成功,但用于病理学细胞核分割的基础模型仍然缺失。初步适应SAM的努力已经取得了一些成功,但并没有提出一个用于多种分割任务的全面模型。为了填补这一空白,我们引入了PathoSAM,这是一个基于多样数据集训练的细胞核分割的VFM。我们的广泛实验表明,它是病理学自动和交互式细胞核实例分割的最新最先进的模型。我们还展示了如何将其适应于其他分割任务,包括语义细胞核分割。对于此任务,我们展示它产生的结果优于流行的方法,但尚未达到当前最先进的CellViT模型的水平。我们的模型是开源的,与流行的数据注释工具兼容。我们还提供了用于全幻灯片图像分割的脚本。我们的代码和模型可在https://github.com/computational-cell-analytics/patho-sam公开访问。
论文及项目相关链接
PDF Published in MIDL 2025
Summary
本文介绍了一种基于训练多样化数据集的Vision Foundation Model(PathoSAM)用于细胞核分割的方法。该方法在数字病理学领域实现了自动和交互式细胞核实例分割的新水平,并可适应其他分割任务。模型开源,兼容数据标注工具,并提供全切片图像分割脚本。
Key Takeaways
- Nucleus segmentation是数字病理学中的重要分析任务,但自动分割方法在新数据上表现不佳,需要用户手动标注细胞核并重新训练特定数据模型。
- Vision Foundation Models(VFMs)如Segment Anything Model(SAM)为自动和交互式分割提供了更稳健的替代方案,但在数字病理学中缺少专门针对细胞核分割的模型。
- PathoSAM是一个基于SAM的VFM模型,经过多样化数据集训练,用于细胞核分割。
- PathoSAM实现了自动和交互式细胞核实例分割的新水平,并可以适应其他分割任务,包括语义细胞核分割。
- 虽然PathoSAM在某些任务上表现出优秀性能,但在某些任务上仍未达到当前最佳模型CellViT的性能。
- PathoSAM模型是开源的,与流行的数据标注工具兼容,并提供全切片图像分割脚本。
点此查看论文截图

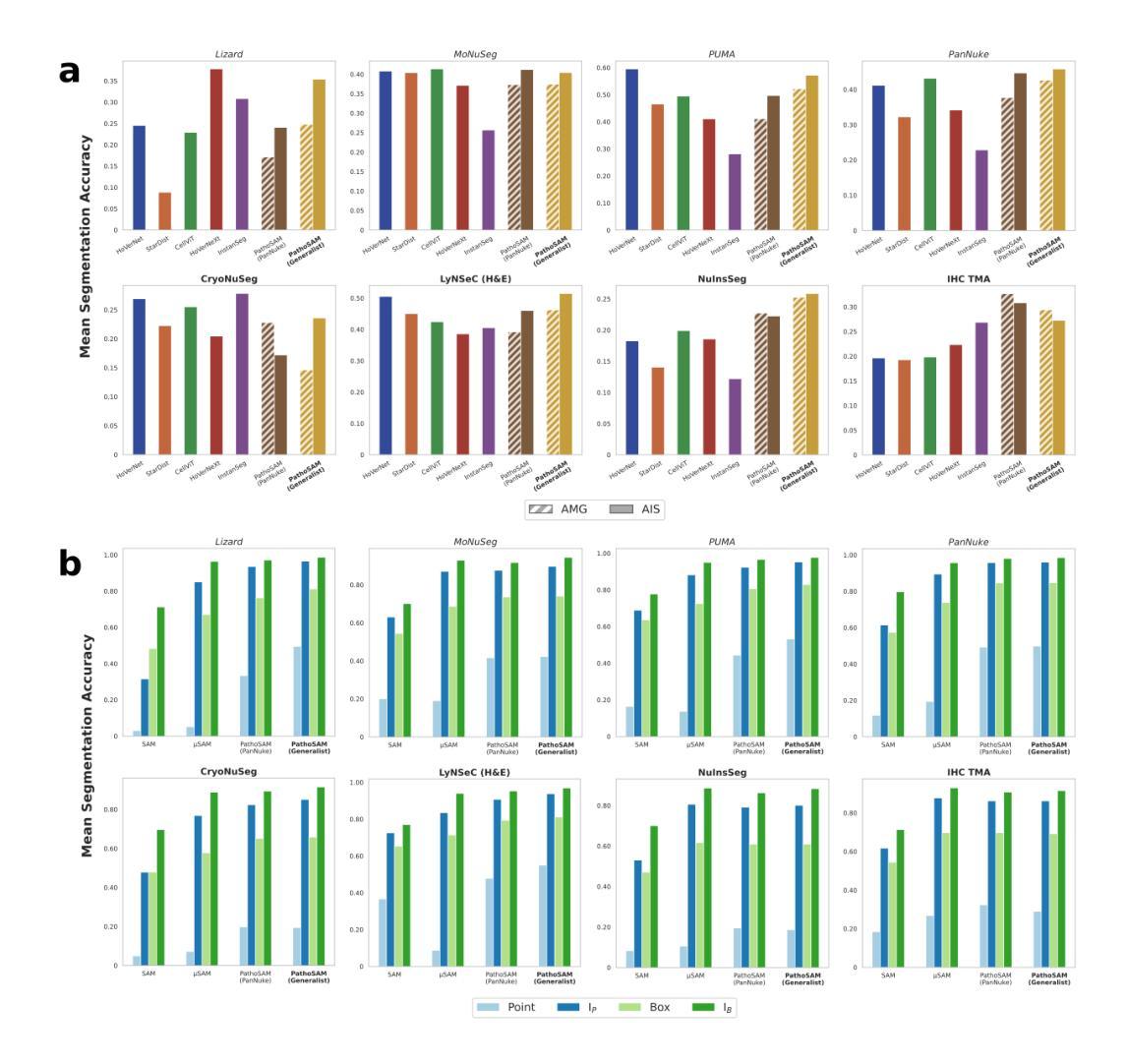
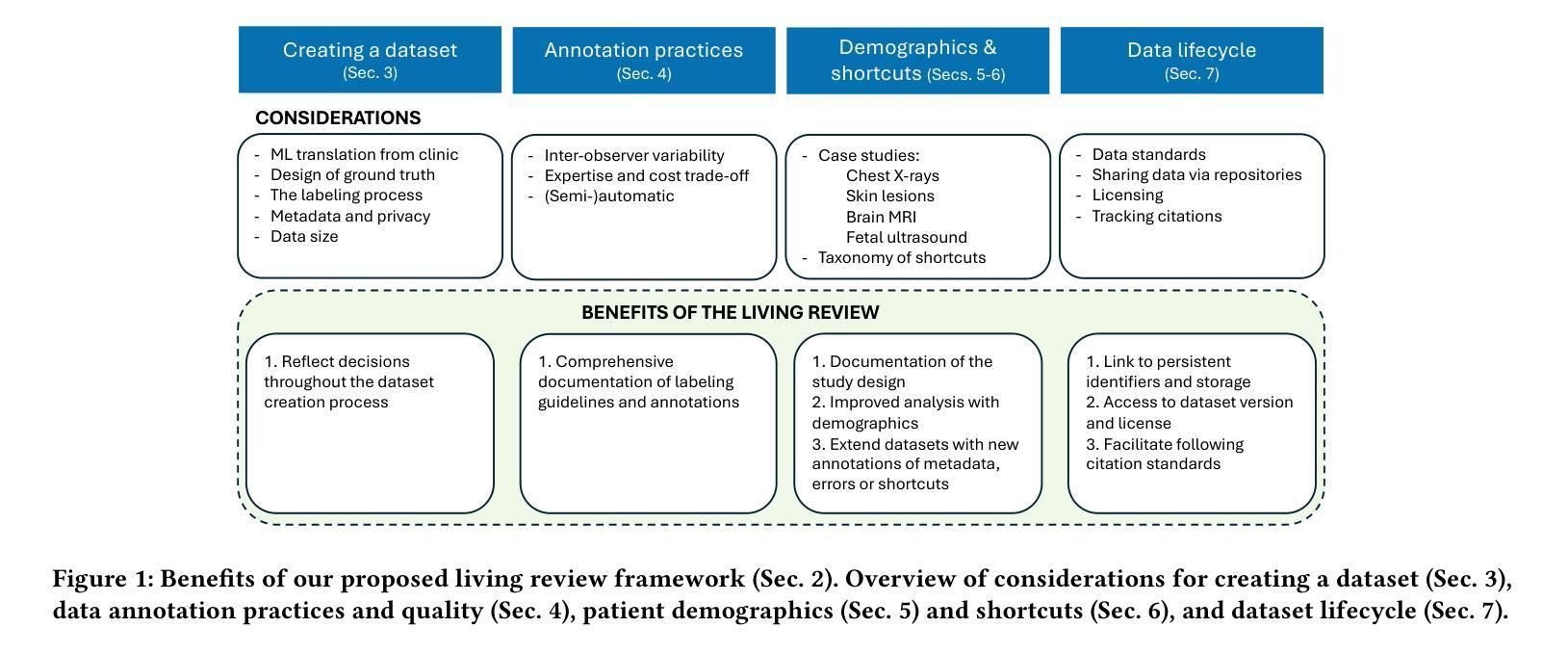
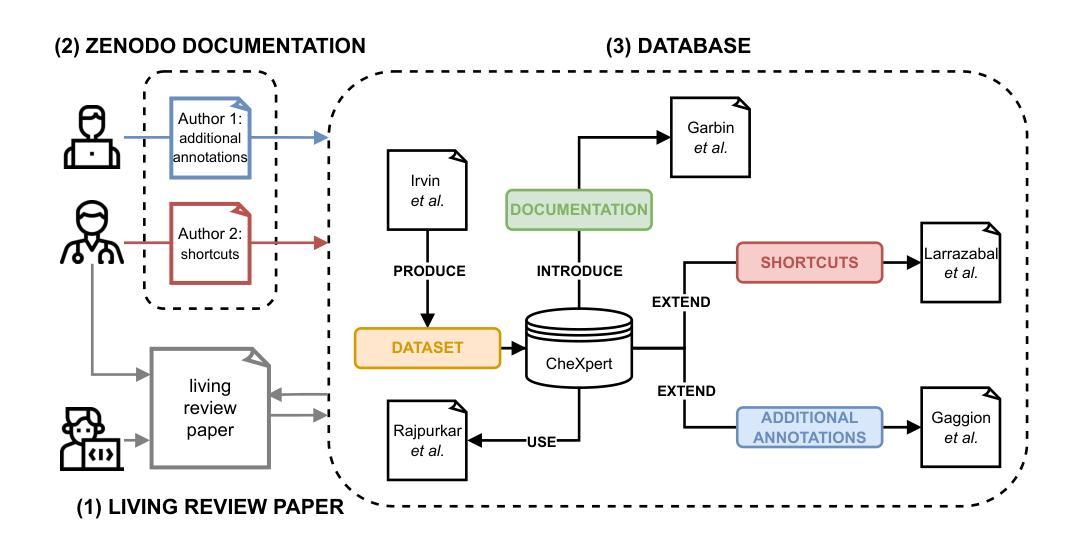
In the Picture: Medical Imaging Datasets, Artifacts, and their Living Review
Authors:Amelia Jiménez-Sánchez, Natalia-Rozalia Avlona, Sarah de Boer, Víctor M. Campello, Aasa Feragen, Enzo Ferrante, Melanie Ganz, Judy Wawira Gichoya, Camila González, Steff Groefsema, Alessa Hering, Adam Hulman, Leo Joskowicz, Dovile Juodelyte, Melih Kandemir, Thijs Kooi, Jorge del Pozo Lérida, Livie Yumeng Li, Andre Pacheco, Tim Rädsch, Mauricio Reyes, Théo Sourget, Bram van Ginneken, David Wen, Nina Weng, Jack Junchi Xu, Hubert Dariusz Zając, Maria A. Zuluaga, Veronika Cheplygina
Datasets play a critical role in medical imaging research, yet issues such as label quality, shortcuts, and metadata are often overlooked. This lack of attention may harm the generalizability of algorithms and, consequently, negatively impact patient outcomes. While existing medical imaging literature reviews mostly focus on machine learning (ML) methods, with only a few focusing on datasets for specific applications, these reviews remain static – they are published once and not updated thereafter. This fails to account for emerging evidence, such as biases, shortcuts, and additional annotations that other researchers may contribute after the dataset is published. We refer to these newly discovered findings of datasets as research artifacts. To address this gap, we propose a living review that continuously tracks public datasets and their associated research artifacts across multiple medical imaging applications. Our approach includes a framework for the living review to monitor data documentation artifacts, and an SQL database to visualize the citation relationships between research artifact and dataset. Lastly, we discuss key considerations for creating medical imaging datasets, review best practices for data annotation, discuss the significance of shortcuts and demographic diversity, and emphasize the importance of managing datasets throughout their entire lifecycle. Our demo is publicly available at http://inthepicture.itu.dk/.
数据集在医学成像研究中扮演着至关重要的角色,然而标签质量、捷径和元数据等问题往往被忽视。这种缺乏关注可能会损害算法的通用性,并因此对患者结果产生负面影响。尽管现有的医学成像文献综述主要集中在机器学习(ML)方法上,只有少数关注特定应用的数据集,但这些综述是静态的——一经发布,就不再更新。这未能考虑到新兴证据,如偏见、捷径和其他研究人员在数据集发布后可能做出的额外注释等。我们将这些新发现的数据集称为研究产物。为了弥补这一空白,我们提出了一种持续追踪多个医学成像应用中的公共数据集及其相关研究产物的动态综述。我们的方法包括一个用于监控数据文档产物和可视化数据集与研究产物之间引文关系的SQL数据库框架。最后,我们讨论了创建医学成像数据集的关键注意事项,回顾了数据注释的最佳实践,讨论了捷径和人口多样性的重要性,并强调了整个数据生命周期中管理数据集的重要性。我们的演示公开访问地址为 http://inthepicture.itu.dk/.
论文及项目相关链接
PDF ACM Conference on Fairness, Accountability, and Transparency - FAccT 2025
Summary
医学成像研究中数据集至关重要,但标签质量、捷径和元数据等问题常被忽视。这些问题可能影响算法泛化能力,进而影响患者治疗效果。现有医学成像文献综述主要关注机器学习(ML)方法,仅少数关注特定应用的数据集,但这些综述一经发布便不再更新,无法涵盖新兴证据,如偏见、捷径和附加注释等研究者贡献的研究工件。为解决此问题,我们提出了一个持续追踪多个医学成像应用公共数据集及其相关研究工件的动态综述。我们的方法包括一个用于监控数据文档工件动态综述的框架和一个SQL数据库,以可视化研究工件与数据集之间的引文关系。最后,我们讨论了创建医学成像数据集的关键考量因素,回顾了数据注释的最佳实践,讨论了捷径和人口多样性的重要性,并强调了整个数据集生命周期中管理数据集的重要性。
Key Takeaways
数据集在医学成像研究中的重要性及其常忽略的问题,如标签质量、捷径和元数据。
现有文献综述的局限性,对新兴证据(如偏见、捷径等研究工件)无法充分覆盖的问题。
提议的“动态综述”的概念来跟踪公共数据集及其相关研究结果工件的应用表现跨多个医学成像领域的观点及其优势。
通过监控数据文档工件的方式提出框架以应对动态综述的要求以及可视化数据库描述研究工件与数据集之间的关系的描述。
讨论创建医学成像数据集时的关键考量因素。
介绍并回顾数据注释的最佳实践及其意义和价值重要性增加新的角度考量对现有的概念有全面更深入的理解对数据和算法有更全面的了解对数据和算法的优化有更清晰的思路对科研研究有更深刻的理解和应用价值对科研实践有重要指导意义强调捷径和人口多样性的重要性在整个数据集中如何避免并有效利用这些捷径增加模型训练过程中的数据多样性的意义对于解决实际应用中的问题提供更全面有效的思路和方向通过理解不同数据和算法的优劣势形成全面的理解和科学的实践建议可以充分利用并规避数据的局限提高研究质量和成果应用的广度对当前学术和实践的影响为进一步的学术研究和技术创新提供了基础和应用价值有助于相关领域的科研工作者更好地理解和应用医学成像数据集。
点此查看论文截图

MADUV: The 1st INTERSPEECH Mice Autism Detection via Ultrasound Vocalization Challenge
Authors:Zijiang Yang, Meishu Song, Xin Jing, Haojie Zhang, Kun Qian, Bin Hu, Kota Tamada, Toru Takumi, Björn W. Schuller, Yoshiharu Yamamoto
The Mice Autism Detection via Ultrasound Vocalization (MADUV) Challenge introduces the first INTERSPEECH challenge focused on detecting autism spectrum disorder (ASD) in mice through their vocalizations. Participants are tasked with developing models to automatically classify mice as either wild-type or ASD models based on recordings with a high sampling rate. Our baseline system employs a simple CNN-based classification using three different spectrogram features. Results demonstrate the feasibility of automated ASD detection, with the considered audible-range features achieving the best performance (UAR of 0.600 for segment-level and 0.625 for subject-level classification). This challenge bridges speech technology and biomedical research, offering opportunities to advance our understanding of ASD models through machine learning approaches. The findings suggest promising directions for vocalization analysis and highlight the potential value of audible and ultrasound vocalizations in ASD detection.
通过超声波发声(MADUV)检测小鼠自闭症(Mice Autism Detection via Ultrasound Vocalization,简称MADUV)挑战引入了首个聚焦于通过小鼠的发声来检测自闭症谱系障碍(ASD)的INTERSPEECH挑战。参赛者的任务是开发模型,根据高采样率的录音自动将小鼠分类为野生型或ASD模型。我们的基线系统采用基于简单卷积神经网络(CNN)的分类方法,使用三种不同的频谱特征。结果表明,自动化检测ASD是可行的,在考虑的音频范围内的特征取得了最佳性能(分段级别的UAR为0.600,主体级别的分类为0.625)。此次挑战将语音技术与生物医学研究相结合,为通过机器学习方法了解ASD模型提供了机会。研究结果表明了发声分析的希望方向,并突出了听觉和超声波发声在ASD检测中的潜在价值。
论文及项目相关链接
PDF 5 pages, 1 figure and 2 tables. Submitted to INTERSPEECH 2025. For MADUV Challenge 2025
Summary
该文本介绍了通过超声发声检测小鼠自闭症(MADUV)挑战,这是首个聚焦于通过小鼠发声检测自闭症谱系障碍(ASD)的INTERSPEECH挑战。参与者需开发模型自动将小鼠分类为野生型或ASD模型,依据的是高采样率录音。基线系统采用简单的基于CNN的分类方法,使用三种不同的频谱特征。结果表明自动化ASD检测的可行性,所考虑的听觉范围特征取得最佳性能(分段级别UAR为0.600,主题级别分类为0.625)。该挑战将语音识别技术与生物医学研究相结合,通过机器学习的方法推动对ASD模型的理解。发现表明声音分析的方向具有希望,并突显了听觉和超声波发声在ASD检测中的潜在价值。
Key Takeaways
- MADUV挑战是首个聚焦于通过小鼠发声检测自闭症谱系障碍(ASD)的INTERSPEECH挑战。
- 参与者需开发模型自动分类小鼠,区分野生型和ASD模型,依据高采样率录音。
- 基线系统采用简单的基于CNN的分类方法,使用三种不同的频谱特征进行分类。
- 自动化ASD检测的可行性得到验证,其中听觉范围特征表现最佳。
- 分段级别和主题级别的分类性能分别达到了UAR 0.600和0.625。
- 该挑战推动了语音识别技术与生物医学研究的结合,通过机器学习的方法加深对ASD模型的理解。
点此查看论文截图

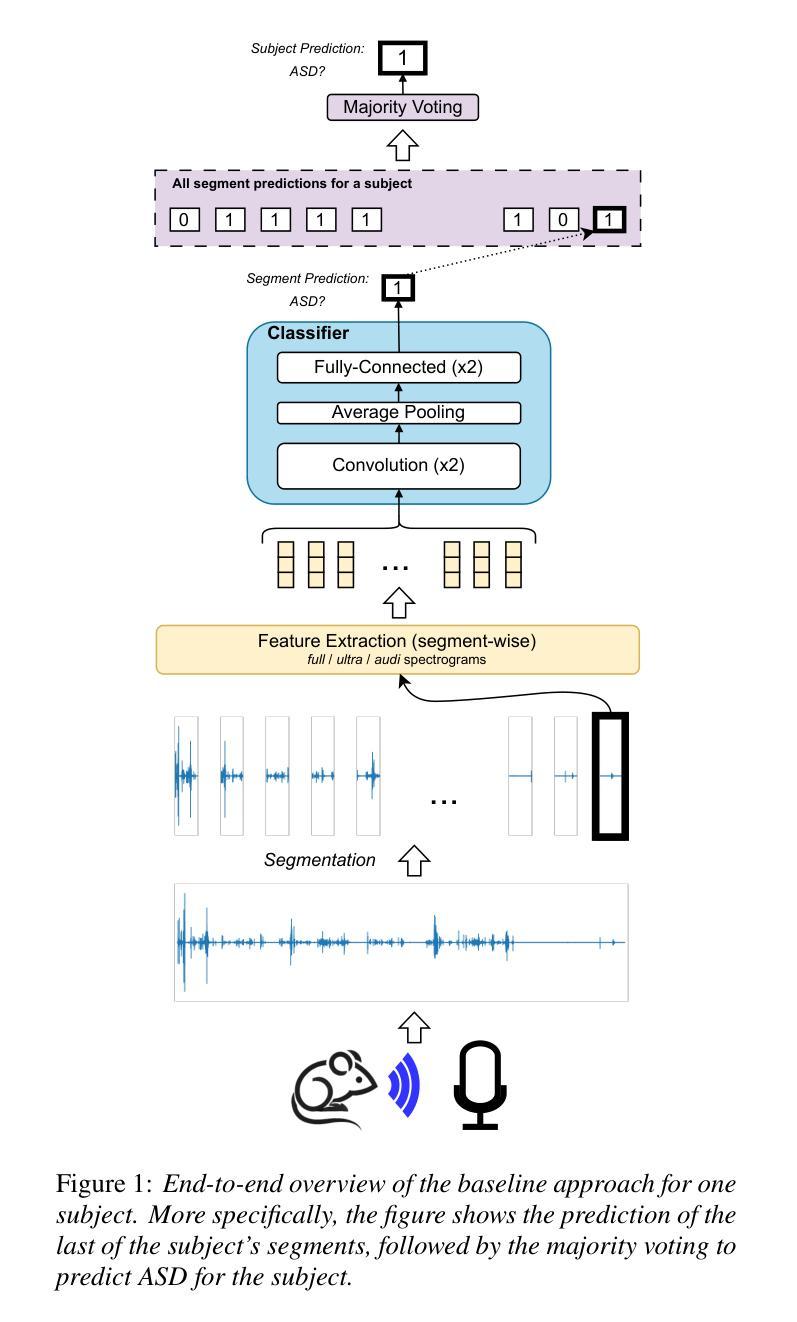

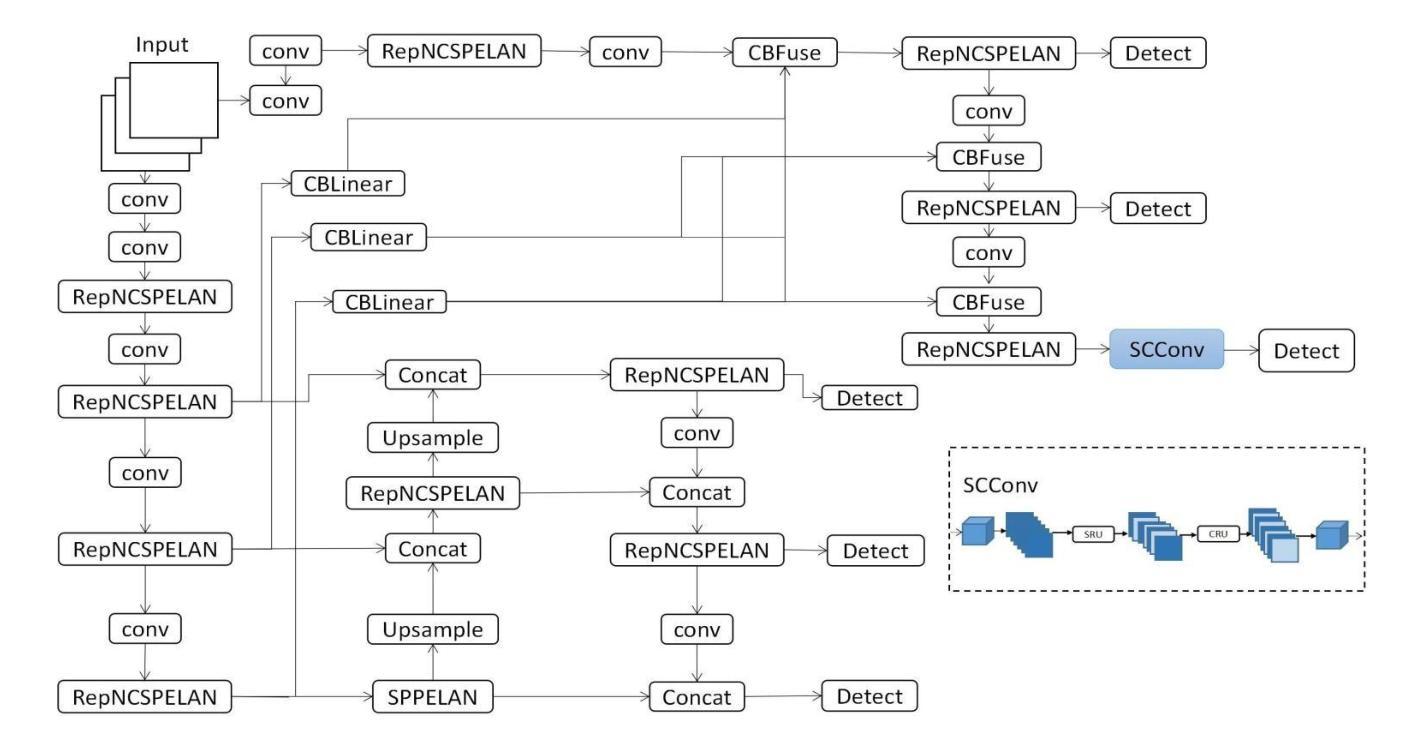
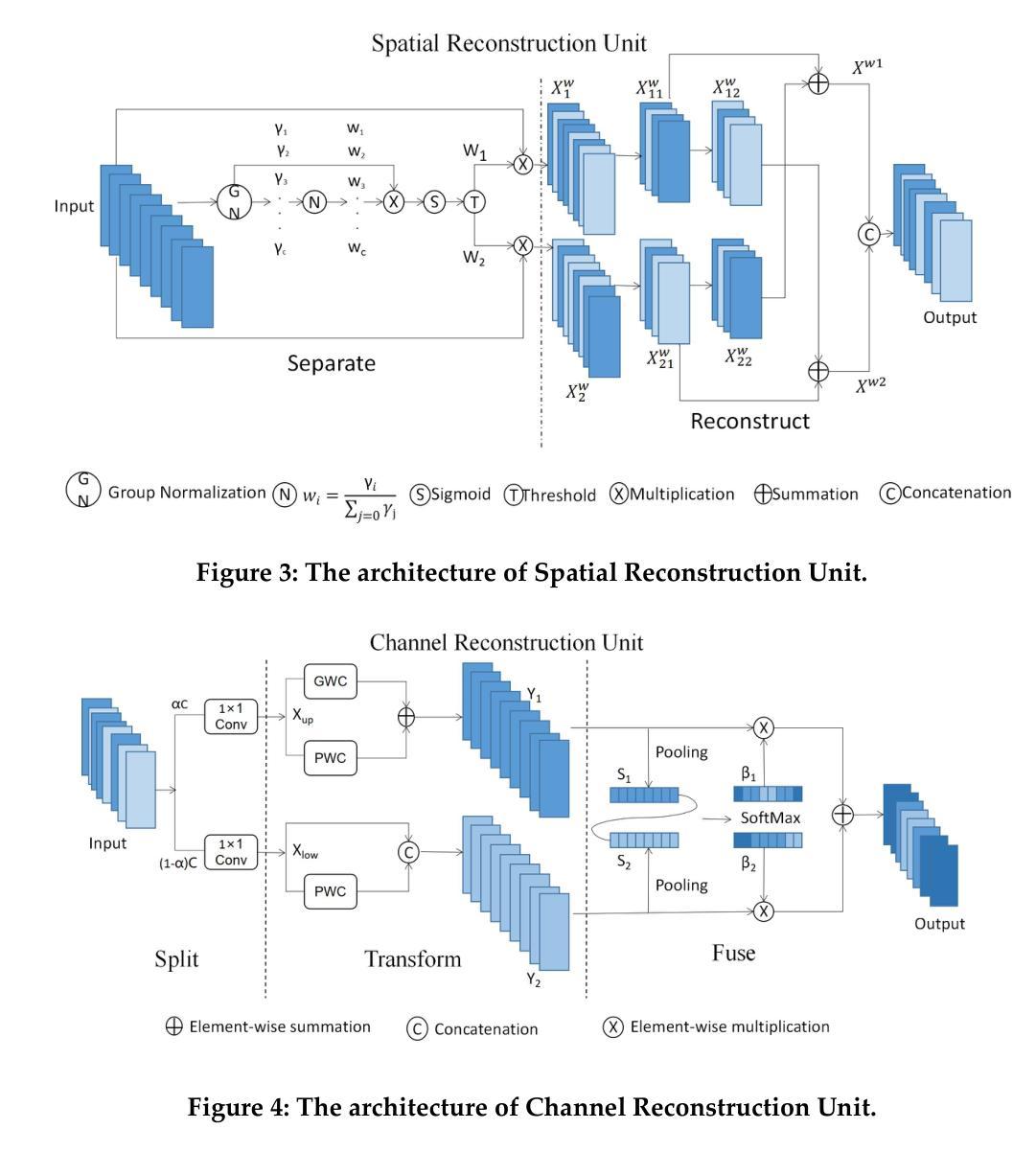
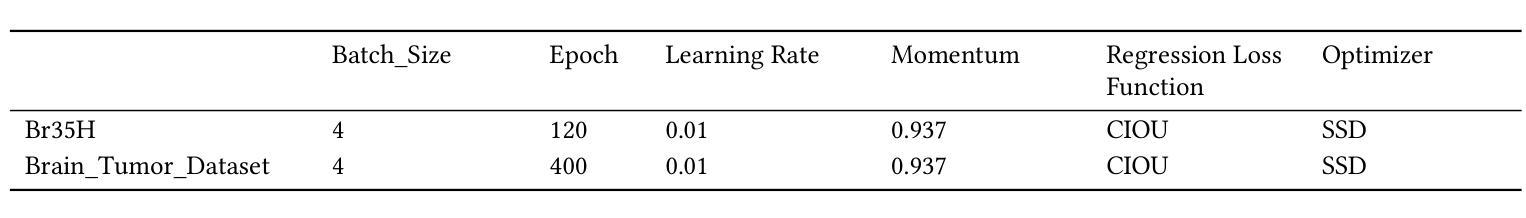
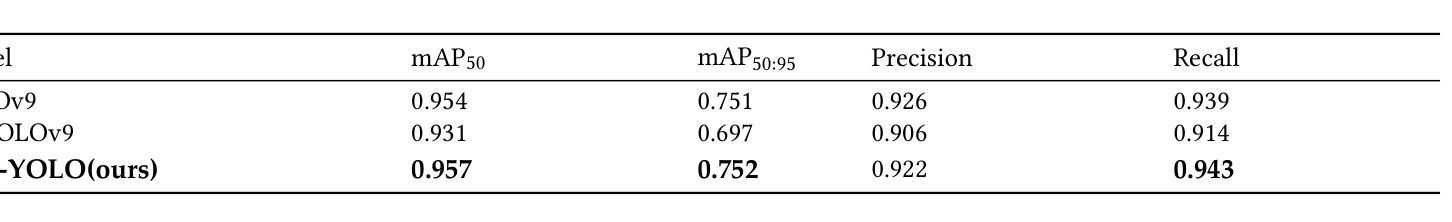
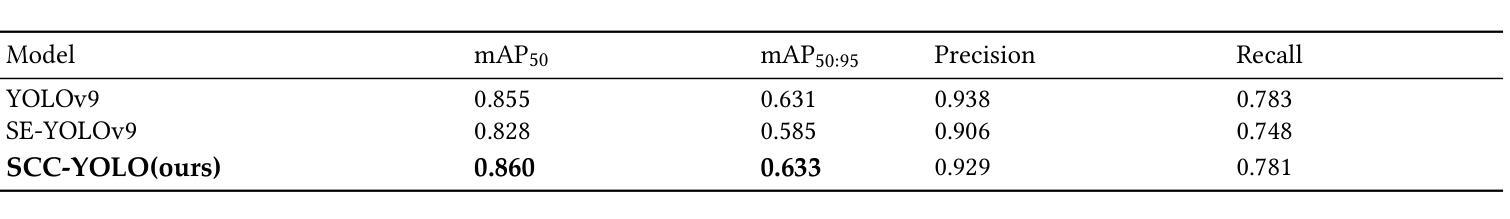
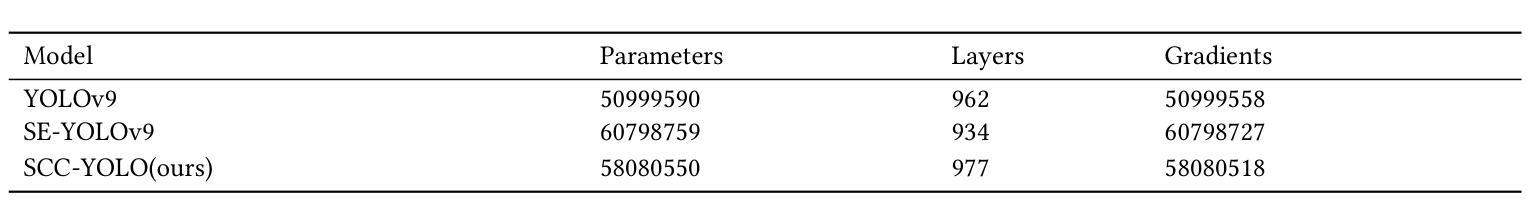
SCC-YOLO: An Improved Object Detector for Assisting in Brain Tumor Diagnosis
Authors:Runci Bai, Guibao Xu, Yanze Shi
Brain tumors can lead to neurological dysfunction, cognitive and psychological changes, increased intracranial pressure, and seizures, posing significant risks to health. The You Only Look Once (YOLO) series has shown superior accuracy in medical imaging object detection. This paper presents a novel SCC-YOLO architecture that integrates the SCConv module into YOLOv9. The SCConv module optimizes convolutional efficiency by reducing spatial and channel redundancy, enhancing image feature learning. We examine the effects of different attention mechanisms with YOLOv9 for brain tumor detection using the Br35H dataset and our custom dataset (Brain_Tumor_Dataset). Results indicate that SCC-YOLO improved mAP50 by 0.3% on the Br35H dataset and by 0.5% on our custom dataset compared to YOLOv9. SCC-YOLO achieves state-of-the-art performance in brain tumor detection.
脑肿瘤可能导致神经功能障碍、认知和心理学变化、颅内压升高和癫痫发作,对健康构成重大风险。You Only Look Once(YOLO)系列已在医学影像目标检测中展现出卓越准确性。本文提出了一种新型的SCC-YOLO架构,它将SCConv模块集成到YOLOv9中。SCConv模块通过减少空间冗余和通道冗余来优化卷积效率,从而增强图像特征学习。我们使用Br35H数据集和我们自己的数据集(Brain_Tumor_Dataset)来检验不同注意力机制对YOLOv9在脑肿瘤检测上的影响。结果表明,与YOLOv9相比,SCC-YOLO在Br35H数据集上的mAP50提高了0.3%,在我们自己的数据集上提高了0.5%。SCC-YOLO在脑肿瘤检测方面达到了最先进的性能。
论文及项目相关链接
Summary
本文介绍了一种新型的SCC-YOLO架构,它将SCConv模块集成到YOLOv9中以提高医学图像中目标检测的准确性。研究结果表明,在脑肿瘤检测方面,SCC-YOLO在Br35H数据集上的mAP50提高了0.3%,在自定义数据集上的mAP50提高了0.5%,达到了当前最先进的性能。
Key Takeaways
- 文中探讨了脑肿瘤可能引发的健康风险,如神经功能障碍、认知和心理变化等。
- YOLO系列在医学成像物体检测中展现出高精度性能。
- 新提出的SCC-YOLO架构集成了SCConv模块以提升图像特征学习能力并优化卷积效率。
- 研究者通过Br35H数据集和自定义数据集(Brain_Tumor_Dataset)评估了不同注意力机制对脑肿瘤检测的影响。
- SCC-YOLO相较于YOLOv9在Br35H数据集上的mAP50提高了0.3%,在自定义数据集上提高了0.5%。
- SCC-YOLO在脑肿瘤检测方面达到了当前最先进的性能水平。
点此查看论文截图

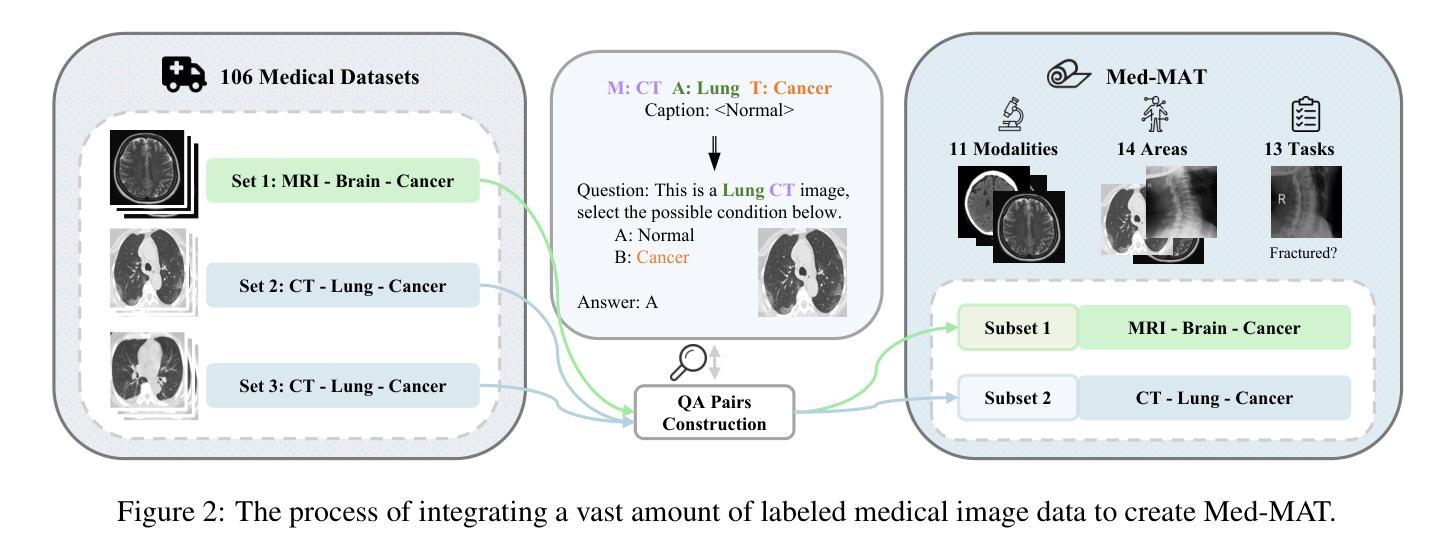
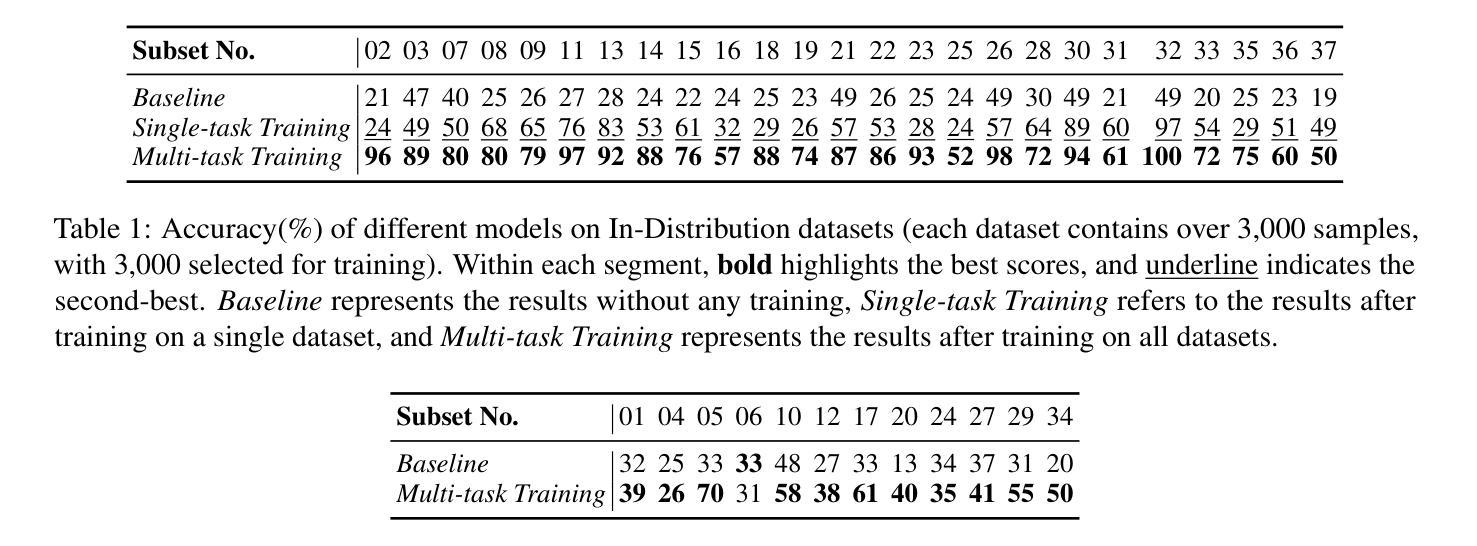
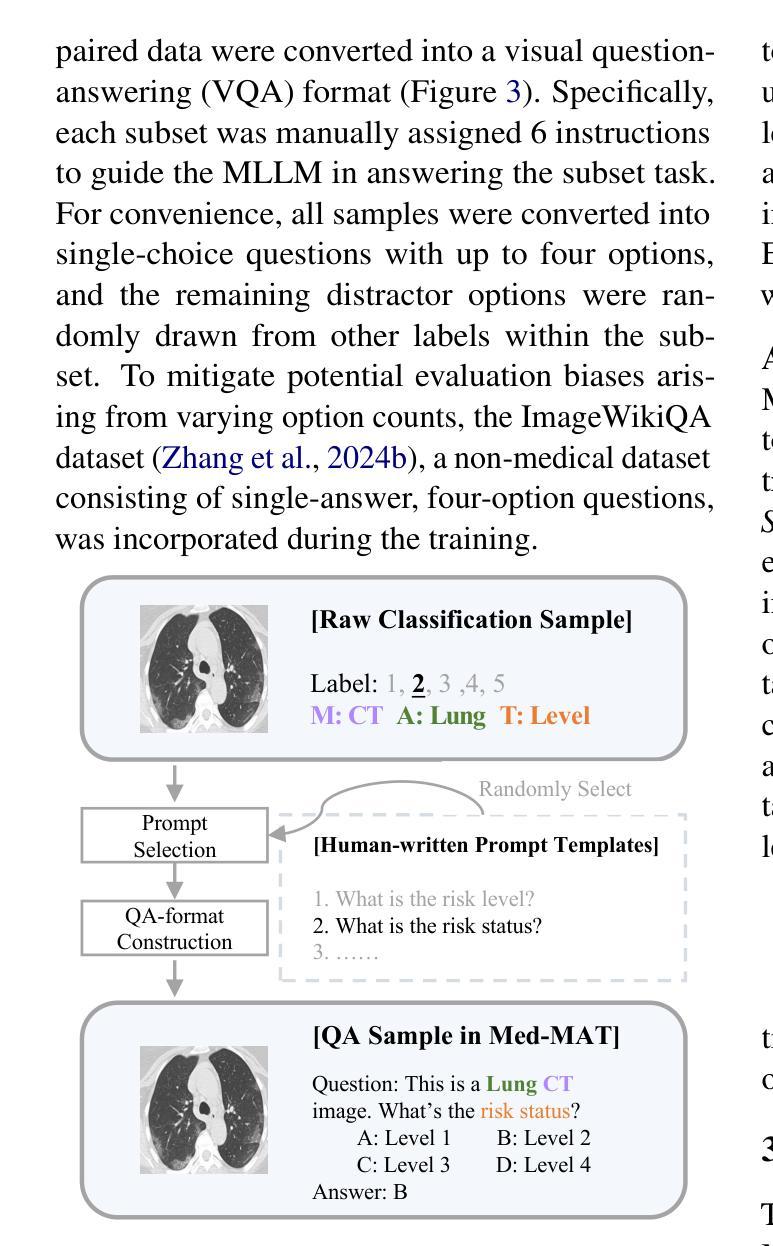
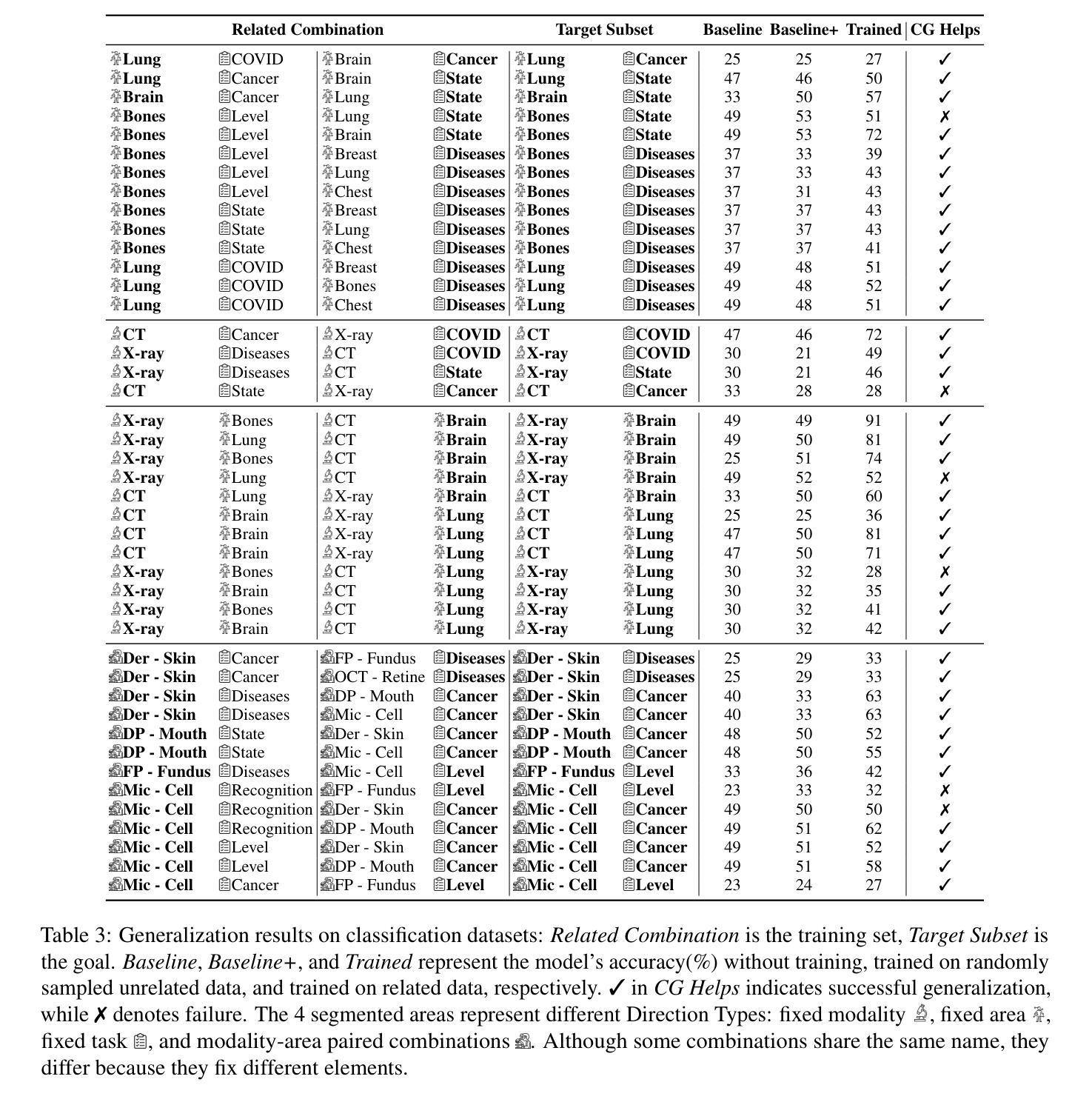
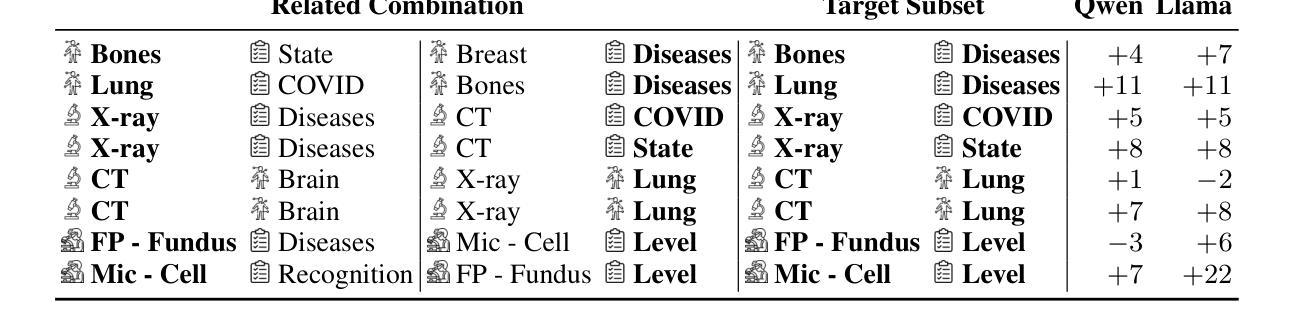
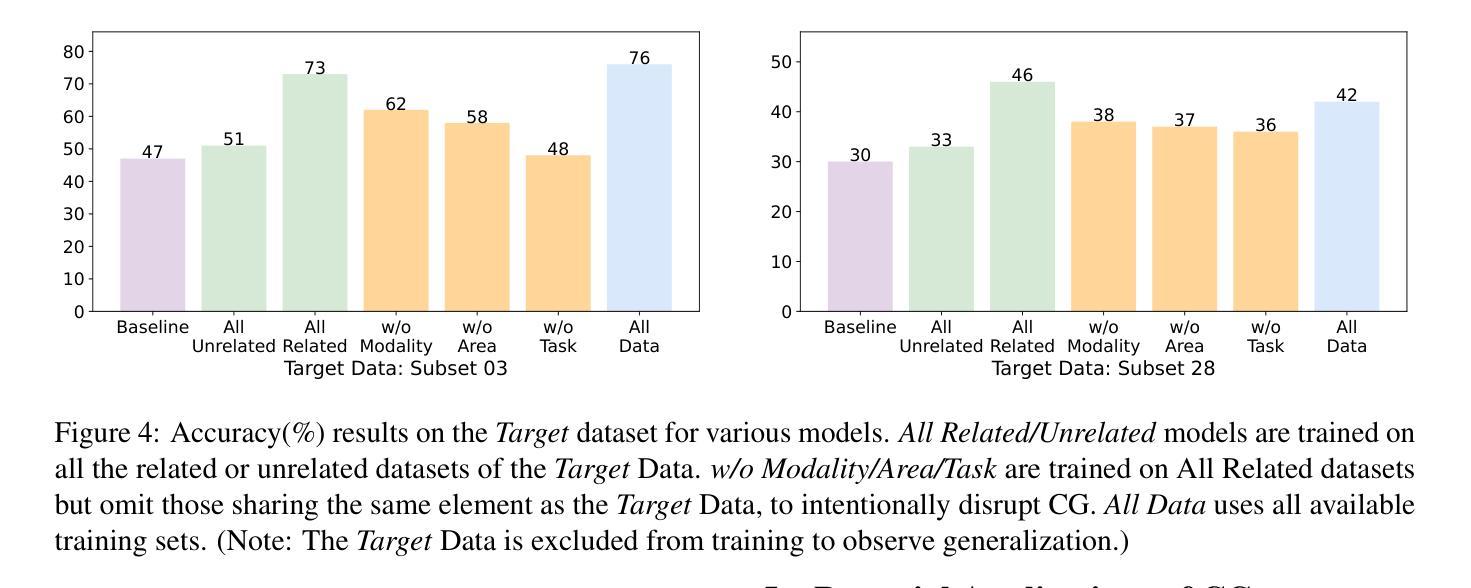
Exploring Compositional Generalization of Multimodal LLMs for Medical Imaging
Authors:Zhenyang Cai, Junying Chen, Rongsheng Wang, Weihong Wang, Yonglin Deng, Dingjie Song, Yize Chen, Zixu Zhang, Benyou Wang
Medical imaging provides essential visual insights for diagnosis, and multimodal large language models (MLLMs) are increasingly utilized for its analysis due to their strong generalization capabilities; however, the underlying factors driving this generalization remain unclear. Current research suggests that multi-task training outperforms single-task as different tasks can benefit each other, but they often overlook the internal relationships within these tasks. To analyze this phenomenon, we attempted to employ compositional generalization (CG), which refers to the models’ ability to understand novel combinations by recombining learned elements, as a guiding framework. Since medical images can be precisely defined by Modality, Anatomical area, and Task, naturally providing an environment for exploring CG, we assembled 106 medical datasets to create Med-MAT for comprehensive experiments. The experiments confirmed that MLLMs can use CG to understand unseen medical images and identified CG as one of the main drivers of the generalization observed in multi-task training. Additionally, further studies demonstrated that CG effectively supports datasets with limited data and confirmed that MLLMs can achieve CG across classification and detection tasks, underscoring its broader generalization potential. Med-MAT is available at https://github.com/FreedomIntelligence/Med-MAT.
医学成像为诊断提供了必要的视觉洞察力,多模态大型语言模型(MLLMs)由于其强大的泛化能力而越来越多地被用于医学图像分析;然而,驱动这种泛化的潜在因素尚不清楚。当前的研究表明,多任务训练优于单任务训练,因为不同的任务可以相互受益,但它们往往会忽略这些任务之间的内部关系。为了分析这一现象,我们尝试采用组合泛化(CG)作为指导框架,组合泛化是指模型通过重新组合已学习的元素来理解新型组合的能力。由于医学图像可以通过模态、解剖部位和任务进行精确定义,自然地为探索CG提供了环境,我们收集了106个医学数据集,创建了Med-MAT用于进行综合实验。实验证实,MLLMs可以利用CG来理解未见过的医学图像,并确定CG是多任务训练中观察到的泛化的主要驱动因素之一。另外,进一步的研究表明,CG有效地支持了数据有限的数据集,并证实MLLMs可以在分类和检测任务中实现CG,突出了其更广泛的泛化潜力。Med-MAT可在https://github.com/FreedomIntelligence/Med-MAT获取。
论文及项目相关链接
Summary
本文探索了医学图像分析中的多模态大型语言模型(MLLMs)的推广能力,尤其是其通过组合学习到的元素理解新颖组合的能力——即组合概括能力(CG)。研究表明,MLLMs可利用CG理解未见过的医学图像,组合概括能力是多任务训练中的主要推广驱动力之一。此外,CG支持有限数据集,并可在分类和检测任务中实现跨任务概括,展现出其更广泛的推广潜力。
Key Takeaways
- 多模态大型语言模型(MLLMs)在医学图像分析中具有强大的推广能力。
- 组合概括能力(CG)是理解新颖组合的关键,有助于模型理解未见过的医学图像。
- 多任务训练在医学图像分析中具有优势,不同任务间可以相互受益。
- CG是多任务训练中的主要推广驱动力之一。
- CG支持有限数据集,显示出其处理资源有限情况的潜力。
- MLLMs能在分类和检测任务中实现跨任务的CG,表明其更广泛的推广潜力。
点此查看论文截图

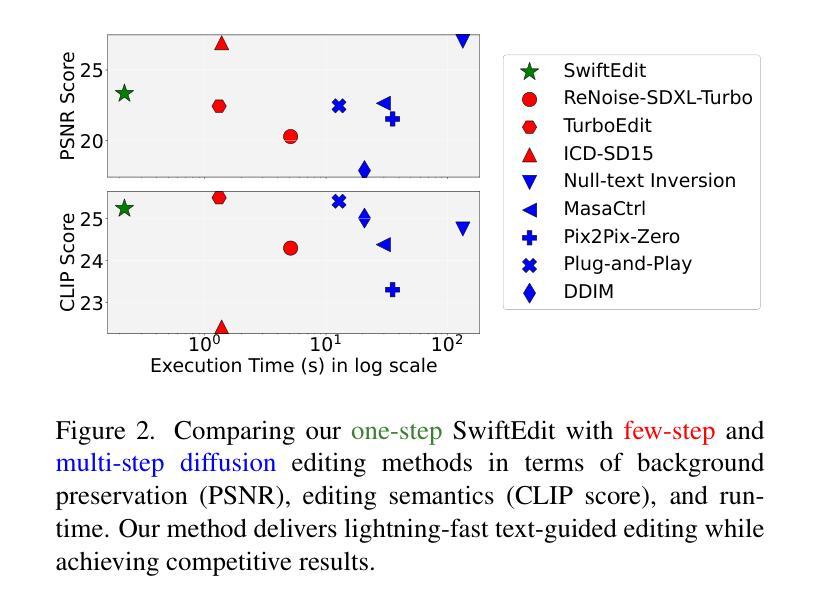
SwiftEdit: Lightning Fast Text-Guided Image Editing via One-Step Diffusion
Authors:Trong-Tung Nguyen, Quang Nguyen, Khoi Nguyen, Anh Tran, Cuong Pham
Recent advances in text-guided image editing enable users to perform image edits through simple text inputs, leveraging the extensive priors of multi-step diffusion-based text-to-image models. However, these methods often fall short of the speed demands required for real-world and on-device applications due to the costly multi-step inversion and sampling process involved. In response to this, we introduce SwiftEdit, a simple yet highly efficient editing tool that achieve instant text-guided image editing (in 0.23s). The advancement of SwiftEdit lies in its two novel contributions: a one-step inversion framework that enables one-step image reconstruction via inversion and a mask-guided editing technique with our proposed attention rescaling mechanism to perform localized image editing. Extensive experiments are provided to demonstrate the effectiveness and efficiency of SwiftEdit. In particular, SwiftEdit enables instant text-guided image editing, which is extremely faster than previous multi-step methods (at least 50 times faster) while maintain a competitive performance in editing results. Our project page is at: https://swift-edit.github.io/
近期文本引导的图像编辑技术取得了进展,使用户能够通过简单的文本输入进行图像编辑,利用多步扩散基础上的文本到图像模型的广泛先验知识。然而,由于涉及昂贵的多步反演和采样过程,这些方法往往难以满足现实世界和在线设备应用的速度要求。对此,我们推出了SwiftEdit,这是一款简单而高效编辑工具,可实现即时文本引导的图像编辑(在0.23秒内)。SwiftEdit的进展在于其两个新颖的贡献:一个一步反演框架,可通过反演实现一步图像重建,以及我们提出的带有注意力调整机制的遮罩引导编辑技术,以执行局部图像编辑。提供了广泛的实验来证明SwiftEdit的有效性和效率。特别是,SwiftEdit能够实现即时文本引导的图像编辑,这比以前的多步方法更快(至少快50倍),同时在编辑结果方面保持竞争力。我们的项目页面是:https://swift-edit.github.io/
简化说明
论文及项目相关链接
PDF 17 pages, 15 figures
Summary
近期文本指导图像编辑技术通过简单的文本输入实现了图像编辑,利用多步扩散基础的文本到图像模型的先验知识。然而,这些方法常常不能满足现实应用和在线设备应用的速度需求,因为它们涉及成本高昂的多步反转和采样过程。为了解决这个问题,我们推出了SwiftEdit,这是一个简单而高效的编辑工具,实现了即时文本指导图像编辑(仅需0.23秒)。SwiftEdit的进步在于其两个新颖的贡献:一步反转框架,可通过反转实现一步图像重建,以及我们提出的注意力重新调整机制的掩膜指导编辑技术,以执行局部图像编辑。
Key Takeaways
- 文本指导图像编辑技术允许通过简单的文本输入进行图像编辑。
- 现有方法因多步反转和采样过程而速度较慢,难以满足实际应用需求。
- SwiftEdit是一个高效编辑工具,实现了即时文本指导图像编辑(0.23秒内)。
- SwiftEdit具有两个新颖贡献:一步反转框架和注意力重新调整机制的掩膜指导编辑技术。
- 一步反转框架可通过反转实现快速图像重建。
- 掩膜指导编辑技术可以执行局部图像编辑。
点此查看论文截图

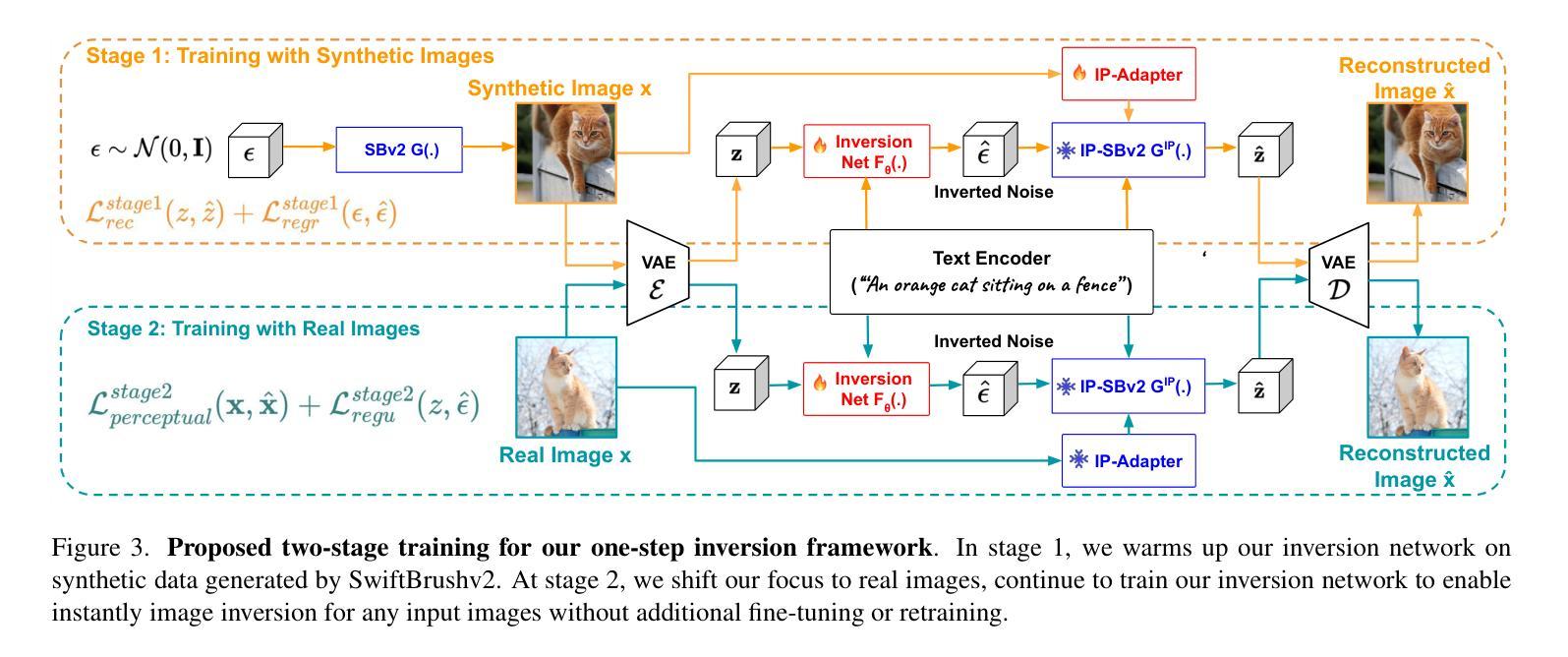
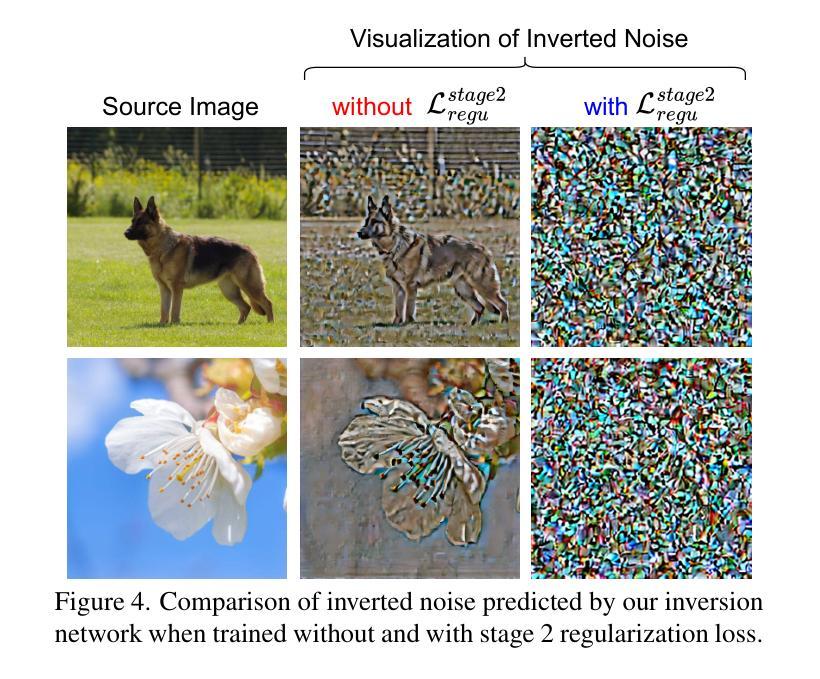
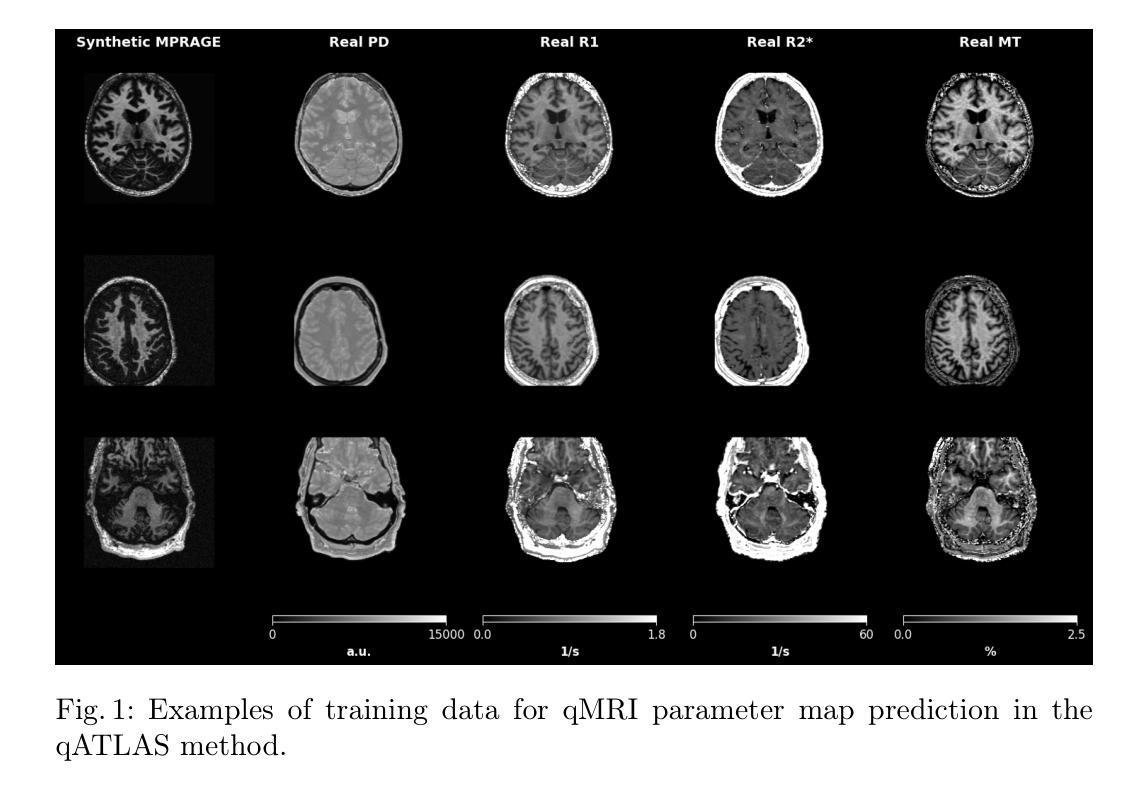
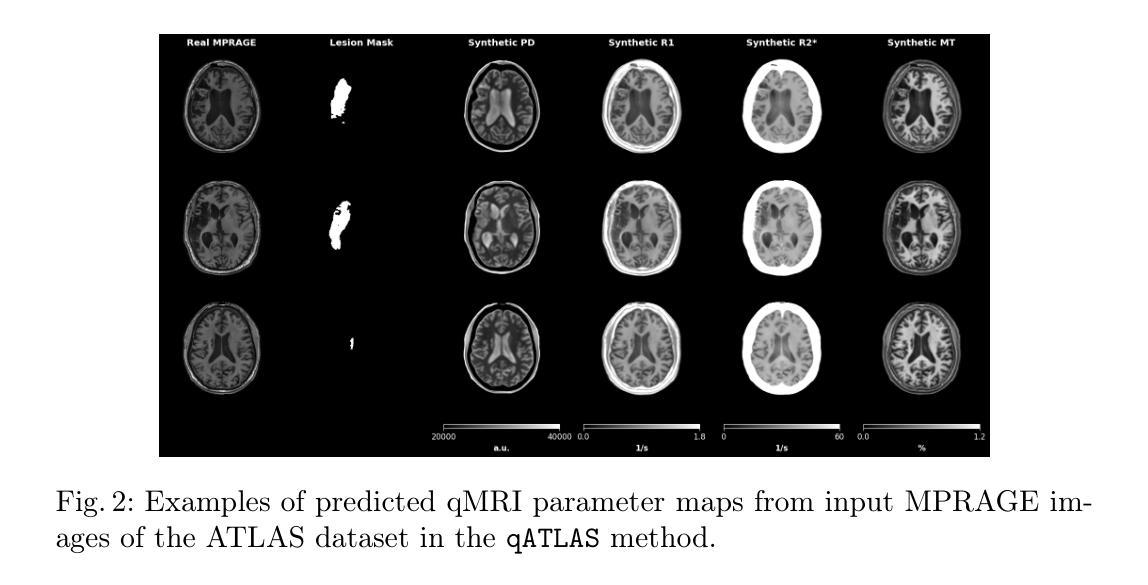
Domain-Agnostic Stroke Lesion Segmentation Using Physics-Constrained Synthetic Data
Authors:Liam Chalcroft, Jenny Crinion, Cathy J. Price, John Ashburner
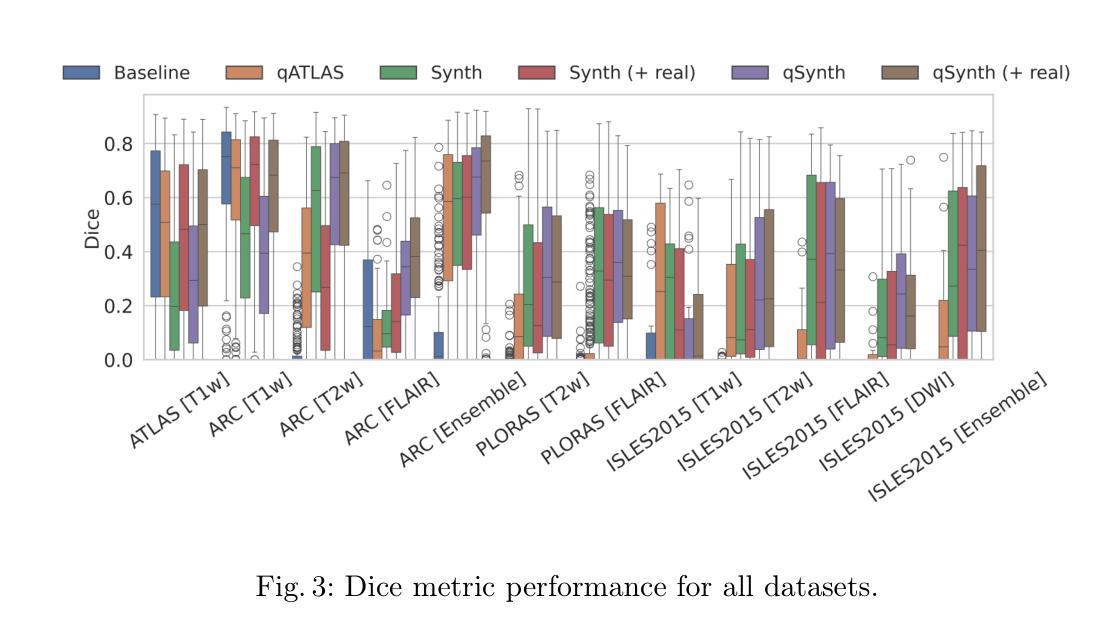
Segmenting stroke lesions in MRI is challenging due to diverse acquisition protocols that limit model generalisability. In this work, we introduce two physics-constrained approaches to generate synthetic quantitative MRI (qMRI) images that improve segmentation robustness across heterogeneous domains. Our first method, $\texttt{qATLAS}$, trains a neural network to estimate qMRI maps from standard MPRAGE images, enabling the simulation of varied MRI sequences with realistic tissue contrasts. The second method, $\texttt{qSynth}$, synthesises qMRI maps directly from tissue labels using label-conditioned Gaussian mixture models, ensuring physical plausibility. Extensive experiments on multiple out-of-domain datasets show that both methods outperform a baseline UNet, with $\texttt{qSynth}$ notably surpassing previous synthetic data approaches. These results highlight the promise of integrating MRI physics into synthetic data generation for robust, generalisable stroke lesion segmentation. Code is available at https://github.com/liamchalcroft/qsynth
在磁共振成像(MRI)中,对卒中病变进行分割是一项挑战,因为不同的采集协议限制了模型的通用性。在这项工作中,我们介绍两种物理约束方法,用于生成合成定量磁共振成像(qMRI)图像,以提高不同异质领域的分割稳健性。我们的第一种方法,
qATLAS,训练神经网络从标准MPRAGE图像估计qMRI地图,能够模拟具有真实组织对比度的各种MRI序列。第二种方法,qSynth,直接从组织标签合成qMRI地图,采用标签条件下的高斯混合模型,确保物理合理性。在多个跨领域数据集上的大量实验表明,这两种方法都优于基线UNet,其中qSynth显著超越了以前的合成数据方法。这些结果突显了将MRI物理学纳入合成数据生成中的潜力,以实现稳健、通用的卒中病变分割。代码可在https://github.com/liamchalcroft/qsynth找到。
论文及项目相关链接
Summary
本文介绍了两种基于物理约束的合成定量核磁共振成像(qMRI)图像生成方法,旨在提高不同异质领域中的分割稳健性。第一种方法qATLAS从标准MPRAGE图像估计qMRI图,模拟多种MRI序列,实现现实组织对比。第二种方法qSynth直接从组织标签合成qMRI图,确保物理合理性。实验表明,两种方法均优于基准UNet,特别是qSynth在合成数据方法上表现出明显优势。集成MRI物理的合成数据生成展现出稳健、通用的脑卒中病灶分割潜力。
Key Takeaways
- 介绍MRI中分割脑卒中病灶的挑战,主要由于采集协议的多样性限制了模型的通用性。
- 提出两种物理约束的合成定量核磁共振成像(qMRI)图像生成方法以提高分割稳健性。
- 第一种方法qATLAS能够从标准MPRAGE图像估计qMRI图,模拟多种MRI序列并实现现实的组织对比。
- 第二种方法qSynth直接从组织标签合成qMRI图,确保合成的图像符合物理规律。
- 在多个跨域数据集上的实验表明,这两种方法均优于基准UNet模型,特别是qSynth表现突出。
- 集成MRI物理的合成数据生成有助于提高模型的稳健性和通用性,在脑卒中病灶分割方面具有潜力。
点此查看论文截图

FactCheXcker: Mitigating Measurement Hallucinations in Chest X-ray Report Generation Models
Authors:Alice Heiman, Xiaoman Zhang, Emma Chen, Sung Eun Kim, Pranav Rajpurkar
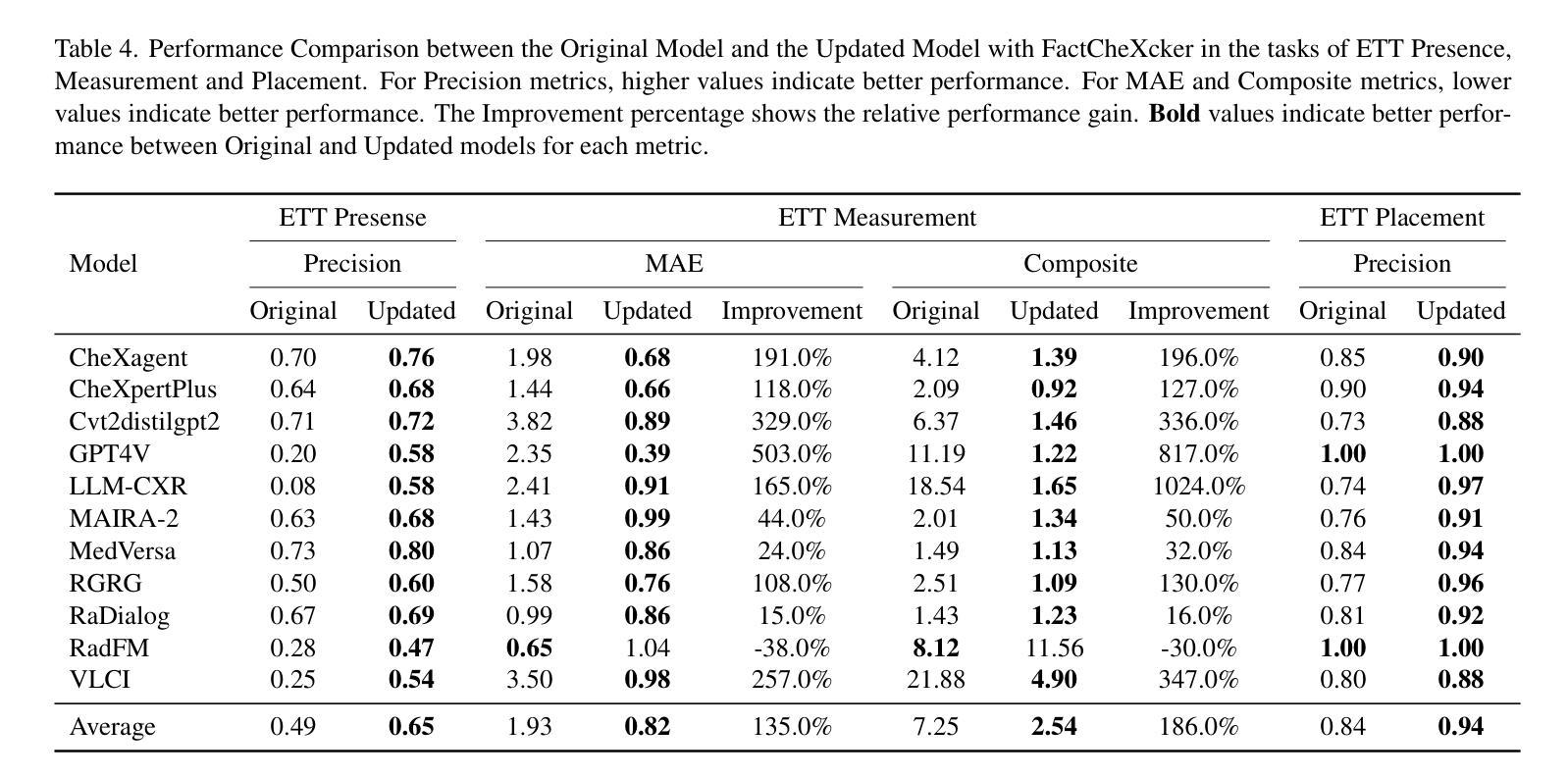
Medical vision-language models often struggle with generating accurate quantitative measurements in radiology reports, leading to hallucinations that undermine clinical reliability. We introduce FactCheXcker, a modular framework that de-hallucinates radiology report measurements by leveraging an improved query-code-update paradigm. Specifically, FactCheXcker employs specialized modules and the code generation capabilities of large language models to solve measurement queries generated based on the original report. After extracting measurable findings, the results are incorporated into an updated report. We evaluate FactCheXcker on endotracheal tube placement, which accounts for an average of 78% of report measurements, using the MIMIC-CXR dataset and 11 medical report-generation models. Our results show that FactCheXcker significantly reduces hallucinations, improves measurement precision, and maintains the quality of the original reports. Specifically, FactCheXcker improves the performance of 10/11 models and achieves an average improvement of 135.0% in reducing measurement hallucinations measured by mean absolute error. Code is available at https://github.com/rajpurkarlab/FactCheXcker.
医学影像语言模型在生成放射学报告中的精确定量测量方面经常遇到困难,这导致出现影响临床可靠性的幻觉。我们引入了FactCheXcker,这是一个模块化框架,它通过改进查询-编码-更新范式来消除放射学报告测量中的幻觉。具体来说,FactCheXcker利用专业模块和大型语言模型的编码生成能力来解决基于原始报告生成的测量查询问题。在提取可测量的发现后,将结果纳入更新的报告中。我们使用MIMIC-CXR数据集和11个医学报告生成模型,对气管导管置入的情况进行了评估,该情况占报告测量的平均77%。我们的结果表明,FactCheXcker显著减少了幻觉,提高了测量精度,并保持了原始报告的质量。具体来说,FactCheXcker提高了其中十个模型的性能,并通过平均绝对误差衡量的测量幻觉减少方面实现了平均135.0%的改进。代码可通过https://github.com/rajpurkarlab/FactCheXcker获取。
论文及项目相关链接
PDF Accepted to CVPR 2025
Summary
医学视觉语言模型在生成放射学报告中的定量测量时常常出现误差,导致产生误导临床的幻觉。我们推出FactCheXcker,一个模块化框架,通过改进查询-编码-更新范式来消除放射学报告中的测量幻觉。具体来说,FactCheXcker利用专业模块和大型语言模型的编码生成能力来解决基于原始报告生成的测量查询问题。在提取出可测量的发现后,将结果纳入更新的报告中。我们在气管插管放置任务上评估了FactCheXcker,该任务占报告测量的平均78%,使用MIMIC-CXR数据集和1 结医学报告生成模型。结果表明,FactCheXcker显著减少了幻觉,提高了测量精度,并保持了原始报告的质量。具体来说,FactCheXcker提高了10/11模型的性能,在平均误差绝对值的测量下,减少测量幻觉的平均改善率为135.0%。代码可在https://github.com/rajpurkarlab/FactCheXcker找到。
Key Takeaways
- 医学视觉语言模型在生成放射学报告中的定量测量时存在误差问题。
- FactCheXcker是一个模块化框架,旨在消除放射学报告中的测量幻觉。
- FactCheXcker通过改进查询-编码-更新范式来解决问题,利用专业模块和大型语言模型的编码生成能力。
- FactCheXcker可以提取并纳入可测量的发现于更新的报告中。
- 在气管插管放置任务上,FactCheXcker显著减少了幻觉并提高了测量精度。
- FactCheXcker对大多数医学报告生成模型都有提高表现的效果。
点此查看论文截图


Leveraging Complementary Attention maps in vision transformers for OCT image analysis
Authors:Haz Sameen Shahgir, Tanjeem Azwad Zaman, Khondker Salman Sayeed, Md. Asif Haider, Sheikh Saifur Rahman Jony, M. Sohel Rahman
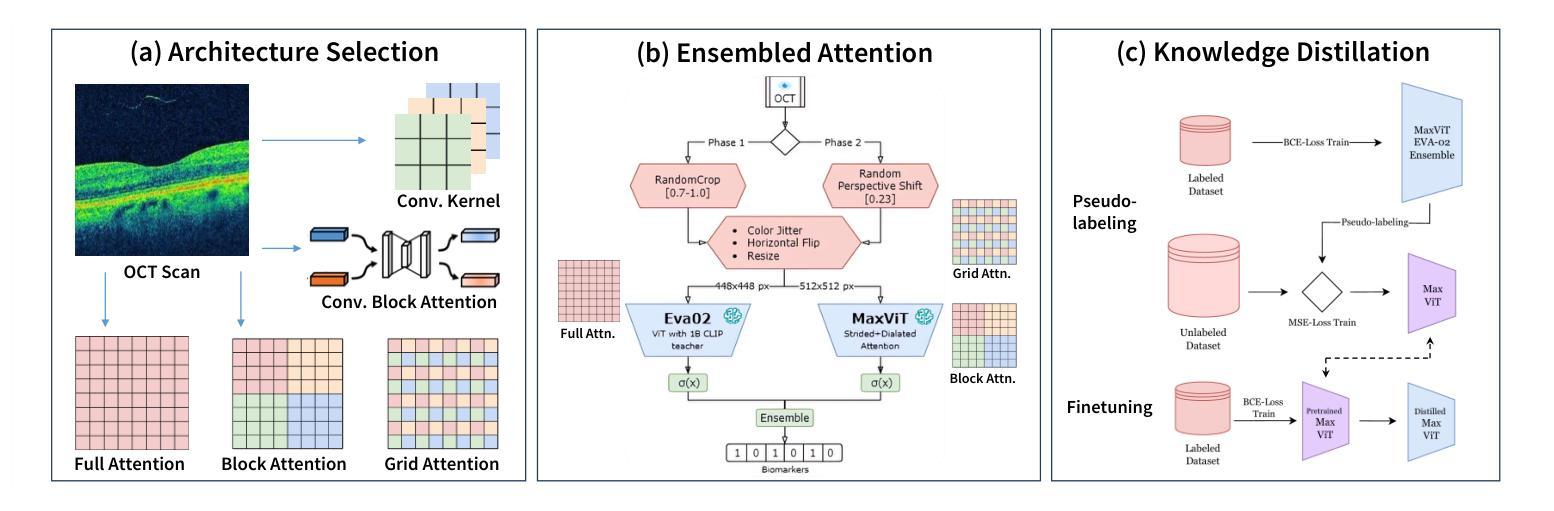
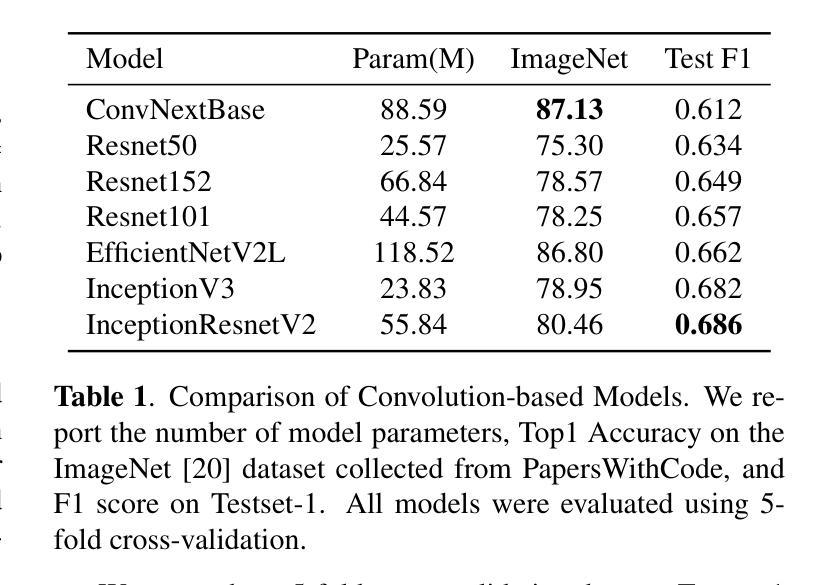
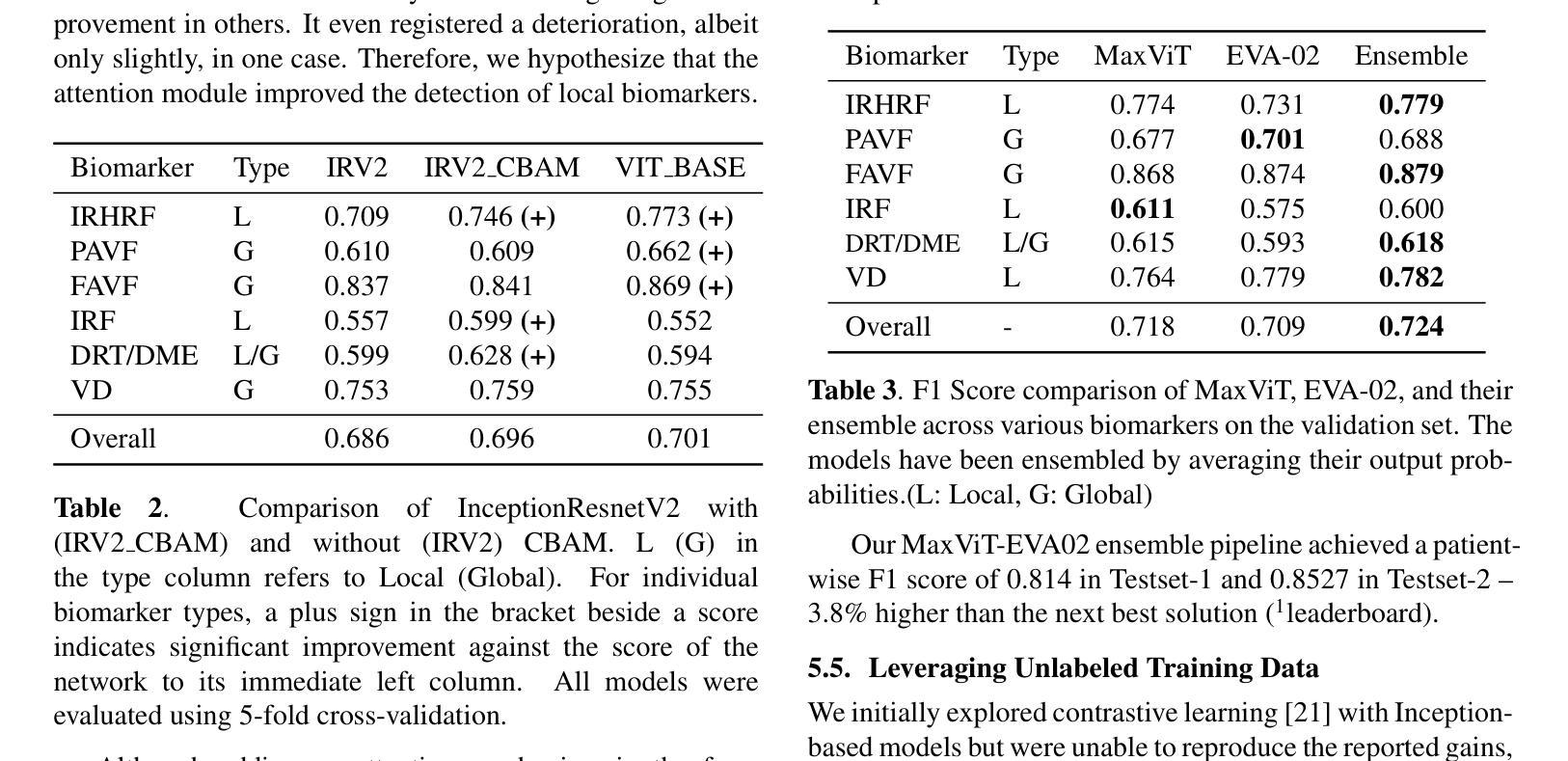
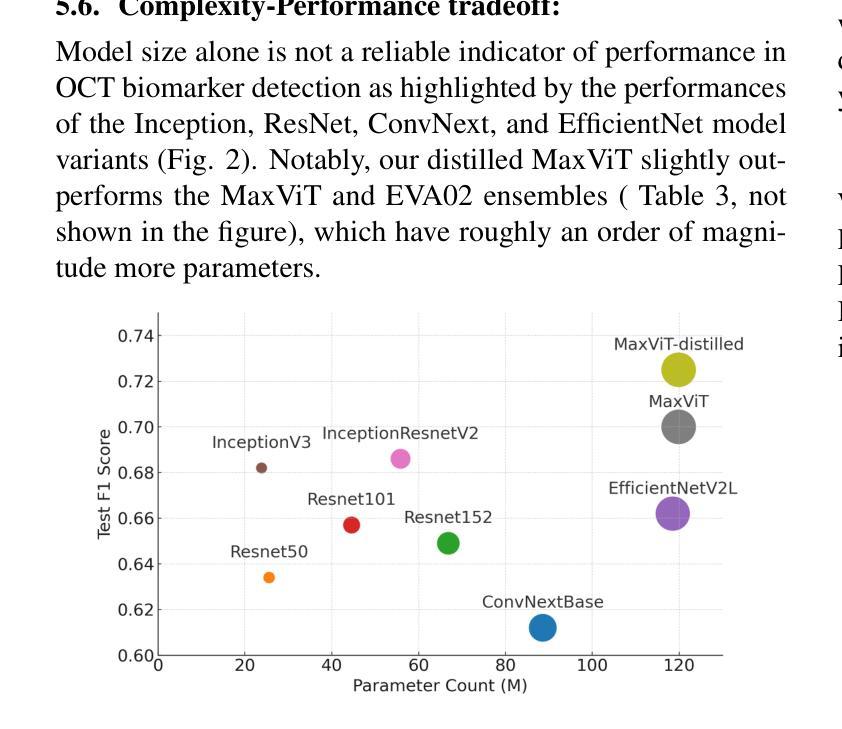
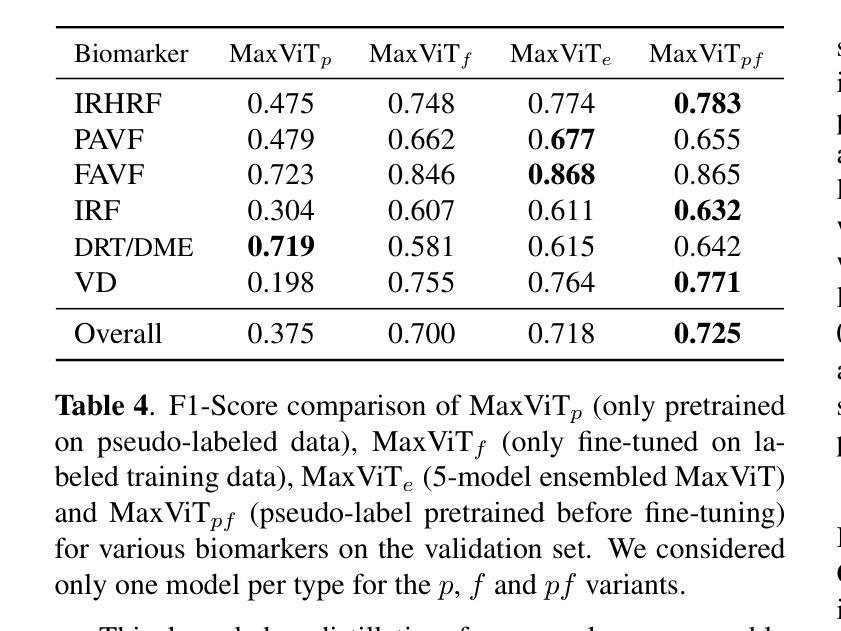
Optical Coherence Tomography (OCT) scan yields all possible cross-section images of a retina for detecting biomarkers linked to optical defects. Due to the high volume of data generated, an automated and reliable biomarker detection pipeline is necessary as a primary screening stage. We outline our new state-of-the-art pipeline for identifying biomarkers from OCT scans. In collaboration with trained ophthalmologists, we identify local and global structures in biomarkers. Through a comprehensive and systematic review of existing vision architectures, we evaluate different convolution and attention mechanisms for biomarker detection. We find that MaxViT, a hybrid vision transformer combining convolution layers with strided attention, is better suited for local feature detection, while EVA-02, a standard vision transformer leveraging pure attention and large-scale knowledge distillation, excels at capturing global features. We ensemble the predictions of both models to achieve first place in the IEEE Video and Image Processing Cup 2023 competition on OCT biomarker detection, achieving a patient-wise F1 score of 0.8527 in the final phase of the competition, scoring 3.8% higher than the next best solution. Finally, we used knowledge distillation to train a single MaxViT to outperform our ensemble at a fraction of the computation cost.
光学相干断层扫描(OCT)扫描可以获取视网膜的所有可能的横截面图像,从而检测与光学缺陷相关的生物标志物。由于产生的大量数据,需要一个自动化和可靠的生物标志物检测流程作为初步筛查阶段。我们概述了我们最新的从OCT扫描中识别生物标志物的先进流程。与训练有素的眼科医生合作,我们识别生物标志物的局部和全局结构。通过对现有视觉架构的全面系统审查,我们评估了用于生物标志物检测的不同卷积和注意力机制。我们发现MaxViT,一种结合卷积层和跨步注意力的混合视觉转换器,更适合于局部特征检测,而EVA-02,一种利用纯注意力和大规模知识蒸馏的标准视觉转换器,擅长捕捉全局特征。我们结合了这两个模型的预测,在IEEE 2023年OCT生物标志物检测比赛中获得第一名,在比赛的最后阶段,患者级别的F1分数达到0.8527,比第二名高出3.8%。最后,我们使用知识蒸馏训练了一个单独的MaxViT,以超过我们组合的表现在很小的计算成本下。
论文及项目相关链接
PDF Accepted in 2025 IEEE International Conference on Image Processing
Summary
本文介绍了一种新型的生物标志物检测管道,该管道结合了光学相干层析扫描技术与先进的视觉技术,旨在从OCT扫描中自动识别和分类与光学缺陷相关的生物标志物。通过与眼科医生的合作,研究人员成功开发了一种集成的预测模型,该模型在IEEE视频和图像处理杯竞赛中取得了第一名,并在最终阶段实现了患者级别的F1分数为0.8527。此外,通过知识蒸馏技术,研究人员还训练了一个单一的MaxViT模型,该模型在计算能力消耗方面表现优于集成模型。
Key Takeaways
- OCT扫描能够生成视网膜的所有可能横截面图像,有助于检测与光学缺陷相关的生物标志物。
- 由于数据量巨大,需要自动化和可靠的生物标志物检测管道作为初步筛选阶段。
- 通过对现有视觉架构的全面系统审查,评估了不同的卷积和注意力机制在生物标志物检测中的应用。
- MaxViT和EVA-02模型分别在局部特征检测和全局特征捕捉方面表现出优势。
- 集成MaxViT和EVA-02模型的预测在IEEE Video and Image Processing Cup 2023竞赛中取得第一名。
- 患者级别的F1分数达到0.8527,较其他最佳解决方案高出3.8%。
点此查看论文截图